IVF算法
🔎 算法原理分步详解
第一阶段:索引构建(建库与分类) 构建索引的目的是为数据建立一个高效的结构化目录,这个过程通常是离线完成的。 1.聚类训练(Clustering) 使用聚类算法(最常用的是 K-Means)将所有向量划分成 nlist个簇(clusters)。nlist是一个关键参数,它决定了空间划分的粒度。每个簇都有一个中心点,称为质心(centroid)。所有这些质心构成了一个“质心表”,相当于图书馆的总分类目录。 2.向量分配(Assignment) 遍历数据集中的每一个向量,计算它与所有质心的距离(如欧氏距离)。将每个向量分配到距离它最近的那个质心所对应的簇中。 3.形成倒排列表(Inverted Lists) 为每一个簇建立一个倒排列表(或称“张贴列表”)。这个列表就像图书馆每个分类书架上的图书清单,它记录了所有属于这个簇的向量的ID以及向量本身(或它的压缩表示)。至此,索引构建完成。
第二阶段:查询处理(快速检索) 当一个新的查询向量到来时,IVF利用已构建好的索引进行快速检索。 1.定位最近簇(Coarse Quantization) 计算查询向量与质心表中所有 nlist个质心的距离。
2.选择候选簇(nprobe 参数控制) 根据上一步的距离结果,选择距离最近的 nprobe个簇作为候选簇。nprobe是IVF算法中最关键的调优参数之一: nprobe越小,搜索范围越小,速度越快,但可能漏掉一些真正近邻(召回率降低)。 nprobe越大,搜索范围越大,召回率越高,但计算量增大,速度变慢。
3.簇内精细比较(Fine Comparison) 在选定的 nprobe个候选簇的倒排列表中,进行精细的距离计算。 具体方式取决于IVF的变体:
- IVF-Flat:直接使用原始的、未压缩的向量与查询向量进行精确距离计算。这种方式精度最高,但内存占用也最大。
- IVF-PQ:为了进一步节省内存和加速计算,会对簇内向量使用乘积量化(Product Quantization) 进行压缩。搜索时使用近似距离计算,这是一种用少量精度换取巨大存储和计算效率提升的策略。
4.结果合并与返回 将所有候选簇中的向量根据与查询向量的距离进行排序,最终返回 Top-K 个最相似的向量作为结果。
VF的性能和效果很大程度上取决于两个核心参数的设置,它们就像这个系统的“调速器”:
- nlist(聚类数):决定了空间的划分粒度。nlist 越大,搜索范围越大,召回率越高;但同时也增加了计算量。
- nprobe(候选簇数):控制了搜索的范围。nprobe 越小,搜索越快,但可能会漏掉一些近邻;nprobe 越大,搜索范围越大,召回率越高,但计算量增大。
IVF算法实现
🧠 IVF算法Python实现
首先,我们导入必要的库:
import numpy as np
from sklearn.metrics.pairwise import cosine_similarity, euclidean_distances
import matplotlib.pyplot as plt
import time
from collections import defaultdict
plt.rcParams['font.sans-serif'] = ['Hiragino Sans GB', 'STHeiti', 'PingFang SC', 'Microsoft YaHei', 'Arial Unicode MS', 'DejaVu Sans']
plt.rcParams['axes.unicode_minus'] = False
# 设置随机种子以保证结果可重现
np.random.seed(42)📊 第一步:生成模拟数据 我们创建一些简单的二维数据,方便可视化理解:
def generate_sample_data(n_samples=1000, dim=2):
"""生成示例数据:三个明显分离的高斯分布簇"""
# 第一个簇
cluster1 = np.random.normal(loc=[2, 2], scale=0.5, size=(n_samples//3, dim))
# 第二个簇
cluster2 = np.random.normal(loc=[8, 3], scale=0.6, size=(n_samples//3, dim))
# 第三个簇
cluster3 = np.random.normal(loc=[5, 8], scale=0.4, size=(n_samples - 2*(n_samples//3), dim))
data = np.vstack([cluster1, cluster2, cluster3])
return data
# 生成数据
data = generate_sample_data()
print(f"数据形状: {data.shape}")输出结果:
数据形状: (1000, 2)⚙️ 第二步:手动实现K-means聚类 这是IVF算法的核心预处理步骤:
class SimpleKMeans:
"""简化的K-means实现用于IVF聚类"""
def __init__(self, n_clusters=3, max_iters=100):
self.n_clusters = n_clusters
self.max_iters = max_iters
self.centroids = None
self.labels_ = None
def fit(self, X):
n_samples, n_features = X.shape
# 1. 随机初始化质心
random_indices = np.random.choice(n_samples, self.n_clusters, replace=False)
self.centroids = X[random_indices]
for iteration in range(self.max_iters):
# 2. 分配每个点到最近的质心
distances = euclidean_distances(X, self.centroids)
labels = np.argmin(distances, axis=1)
# 3. 更新质心位置
new_centroids = np.array([X[labels == i].mean(axis=0) for i in range(self.n_clusters)])
# 检查收敛
if np.allclose(self.centroids, new_centroids):
break
self.centroids = new_centroids
self.labels_ = labels
return self📁 第三步:实现倒排文件索引(IVF) 现在实现完整的IVF索引结构:
class SimpleIVF:
"""简化的IVF实现"""
def __init__(self, n_clusters=3, n_probe=2):
self.n_clusters = n_clusters
self.n_probe = n_probe # 搜索时探测的簇数量
self.kmeans = None
self.inverted_lists = None # 倒排列表
self.centroids = None
self.is_trained = False
def train(self, data):
"""训练IVF索引:对数据进行聚类"""
print("开始训练IVF索引...")
self.kmeans = SimpleKMeans(n_clusters=self.n_clusters)
self.kmeans.fit(data)
self.centroids = self.kmeans.centroids
self.is_trained = True
print(f"训练完成,得到{self.n_clusters}个簇")
def build_index(self, data):
"""构建倒排索引"""
if not self.is_trained:
self.train(data)
# 初始化倒排列表
self.inverted_lists = defaultdict(list)
# 将每个向量分配到最近的簇
distances = euclidean_distances(data, self.centroids)
labels = np.argmin(distances, axis=1)
# 构建倒排列表:簇ID -> 该簇中所有向量的索引
for idx, label in enumerate(labels):
self.inverted_lists[label].append(idx)
print("倒排索引构建完成:")
for cluster_id, items in self.inverted_lists.items():
print(f" 簇{cluster_id}: {len(items)}个向量")
def search(self, query, k=5, data=None):
"""IVF搜索:先找最近的簇,然后在簇内搜索"""
if data is None:
data = self.data
# 1. 粗略搜索:找到最近的n_probe个簇
distances_to_centroids = euclidean_distances([query], self.centroids)[0]
nearest_cluster_indices = np.argsort(distances_to_centroids)[:self.n_probe]
# 2. 精细搜索:在选中的簇内进行暴力搜索
candidate_indices = []
for cluster_idx in nearest_cluster_indices:
candidate_indices.extend(self.inverted_lists[cluster_idx])
if not candidate_indices:
return [], []
# 在候选向量中计算距离
candidate_vectors = data[candidate_indices]
distances = euclidean_distances([query], candidate_vectors)[0]
# 获取最近的k个结果
if k > len(distances):
k = len(distances)
nearest_indices_within_candidates = np.argsort(distances)[:k]
# 映射回原始索引
final_indices = [candidate_indices[i] for i in nearest_indices_within_candidates]
final_distances = distances[nearest_indices_within_candidates]
return final_indices, final_distances
def brute_force_search(self, query, k=5, data=None):
"""暴力搜索作为对比基准"""
if data is None:
data = self.data
distances = euclidean_distances([query], data)[0]
nearest_indices = np.argsort(distances)[:k]
return nearest_indices, distances[nearest_indices]🔍 第四步:算法实现 让我们用一个完整的例子来展示IVF的工作原理:
def demonstrate_ivf():
"""完整演示IVF算法"""
print("=" * 60)
print("IVF算法演示")
print("=" * 60)
# 1. 生成数据
data = generate_sample_data(300, 2)
print(f"生成{len(data)}个二维数据点")
# 2. 创建并训练IVF索引
ivf = SimpleIVF(n_clusters=3, n_probe=2)
ivf.data = data # 保存数据引用
ivf.build_index(data)
# 3. 选择一个查询点
query_point = np.array([5.0, 5.0])
print(f"\n查询点: {query_point}")
# 4. 使用IVF搜索
start_time = time.time()
ivf_indices, ivf_distances = ivf.search(query_point, k=5, data=data)
ivf_time = time.time() - start_time
# 5. 使用暴力搜索作为对比
start_time = time.time()
bf_indices, bf_distances = ivf.brute_force_search(query_point, k=5, data=data)
bf_time = time.time() - start_time
# 6. 显示结果
print(f"\n搜索结果对比:")
print(f"IVF搜索 - 找到{len(ivf_indices)}个最近邻, 耗时: {ivf_time:.6f}秒")
print(f"暴力搜索 - 找到{len(bf_indices)}个最近邻, 耗时: {bf_time:.6f}秒")
print(f"\n速度提升: {bf_time/ivf_time:.2f}倍")
print(f"\n最近邻索引 (IVF): {ivf_indices}")
print(f"最近邻索引 (暴力): {bf_indices}")
# 7. 检查召回率
intersection = set(ivf_indices) & set(bf_indices)
recall = len(intersection) / len(bf_indices)
print(f"召回率: {recall:.2%} ({len(intersection)}/{len(bf_indices)})")
return ivf, data, query_point, ivf_indices, bf_indices
# 运行演示
ivf, data, query, ivf_results, bf_results = demonstrate_ivf()结果输出:
============================================================
IVF算法演示
============================================================
生成: 50,000个二维数据点
IVF参数配置:
聚类数量 (n_clusters): 111
搜索簇数 (n_probe): 13
开始训练IVF索引...
训练完成,得到111个簇
倒排索引构建完成「展示前5个簇」:
簇23: 314个向量
簇43: 348个向量
簇59: 166个向量
簇6: 687个向量
簇32: 682个向量
查询点: [5 5]
搜索结果对比:
IVF搜索 - 找到10个最近邻, 耗时: 0.001200秒
暴力搜索 - 找到10个最近邻, 耗时: 0.004839秒
速度提升: 4.03倍
搜索比例: 10.0% (5,011/50,000)
最近邻索引 (IVF): [26738, 18695, 49731, 35576, 47988, 31669, 44706, 41498, 35176, 44646]
最近邻索引 (暴力): [26738 18695 49731 35576 47988 31669 44706 41498 35176 44646]
召回率: 100.0% (10/10)📈 第五步:可视化结果 让我们用图形化的方式展示IVF的工作原理:
def visualize_ivf(ivf, data, query, ivf_results, bf_results):
"""IVF算法可视化"""
plt.figure(figsize=(15, 10))
colors = ['#FF6B6B', '#4ECDC4', '#45B7D1', '#96CEB4', '#FFEAA7', '#DDA0DD']
# 计算查询相关信息
distances_to_centroids = euclidean_distances([query], ivf.centroids)[0]
nearest_cluster_indices = np.argsort(distances_to_centroids)[:ivf.n_probe]
# 1. 原始数据分布
plt.subplot(2, 3, 1)
plt.scatter(data[:, 0], data[:, 1], c='lightblue', alpha=0.6, s=20, label='数据点')
plt.title('1. 原始数据分布', fontweight='bold')
plt.legend(fontsize=8)
plt.grid(True, alpha=0.3)
# 2. K-means聚类
plt.subplot(2, 3, 2)
for cluster_id, indices in ivf.inverted_lists.items():
cluster_data = data[indices]
plt.scatter(cluster_data[:, 0], cluster_data[:, 1],
c=colors[cluster_id % len(colors)], alpha=0.7, s=25,
label=f'簇{cluster_id}')
plt.scatter(ivf.centroids[:, 0], ivf.centroids[:, 1],
c='black', marker='X', s=100, linewidths=2, label='质心')
plt.title('2. K-means聚类', fontweight='bold')
plt.legend(fontsize=8)
plt.grid(True, alpha=0.3)
# 3. 粗略搜索
plt.subplot(2, 3, 3)
# 背景:显示所有数据点(淡化)
plt.scatter(data[:, 0], data[:, 1], c='lightgray', alpha=0.2, s=10)
# 显示所有质心,用不同透明度区分选中和未选中
selected_centroids = set(nearest_cluster_indices)
for i, centroid in enumerate(ivf.centroids):
if i in selected_centroids:
# 选中的质心:高亮显示
plt.scatter(centroid[0], centroid[1], c='red', marker='X',
s=120, linewidths=2, edgecolors='darkred', alpha=0.9)
# 显示到查询点的距离线
plt.plot([query[0], centroid[0]], [query[1], centroid[1]],
'r-', alpha=0.8, linewidth=2)
# 标注距离值
dist = np.linalg.norm(query - centroid)
mid_x, mid_y = (query[0] + centroid[0])/2, (query[1] + centroid[1])/2
plt.annotate(f'{dist:.1f}', (mid_x, mid_y),
xytext=(0, 10), textcoords='offset points',
fontsize=8, ha='center', color='red', fontweight='bold',
bbox=dict(boxstyle='round,pad=0.2', facecolor='white', alpha=0.8))
else:
# 未选中的质心:淡化显示
plt.scatter(centroid[0], centroid[1], c='gray', marker='X',
s=80, alpha=0.4, linewidths=1)
# 显示到查询点的距离线(虚线)
plt.plot([query[0], centroid[0]], [query[1], centroid[1]],
'gray', linestyle=':', alpha=0.3, linewidth=1)
# 查询点
plt.scatter(query[0], query[1], c='#FFD93D', marker='*', s=200,
edgecolors='black', linewidth=2, label='查询点', zorder=10)
# 添加选择说明
plt.title(f'3. 粗略搜索:选择最近的{ivf.n_probe}个质心', fontweight='bold')
# 创建图例
from matplotlib.lines import Line2D
legend_elements = [
Line2D([0], [0], marker='*', color='w', markerfacecolor='#FFD93D',
markersize=12, markeredgecolor='black', label='查询点'),
Line2D([0], [0], marker='X', color='w', markerfacecolor='red',
markersize=10, markeredgecolor='darkred', label='选中质心'),
Line2D([0], [0], marker='X', color='w', markerfacecolor='gray',
markersize=8, alpha=0.6, label='未选中质心'),
Line2D([0], [0], color='red', linewidth=2, label='选中距离'),
Line2D([0], [0], color='gray', linestyle=':', alpha=0.5, label='未选中距离')
]
plt.legend(handles=legend_elements, fontsize=7, loc='upper right')
plt.grid(True, alpha=0.3)
# 4. 精细搜索
plt.subplot(2, 3, 4)
# 背景:显示所有数据点(淡化)
plt.scatter(data[:, 0], data[:, 1], c='lightgray', alpha=0.2, s=10)
# 显示选中的质心
for cluster_idx in nearest_cluster_indices:
plt.scatter(ivf.centroids[cluster_idx, 0], ivf.centroids[cluster_idx, 1],
c='red', marker='X', s=100, linewidths=2, edgecolors='darkred', alpha=0.8)
# 收集并按簇显示候选向量
candidate_indices = []
cluster_colors = ['#FF6B6B', '#4ECDC4', '#45B7D1', '#96CEB4', '#FFEAA7']
for i, cluster_idx in enumerate(nearest_cluster_indices):
cluster_candidates = ivf.inverted_lists[cluster_idx]
candidate_indices.extend(cluster_candidates)
# 用不同颜色显示不同簇的候选向量
cluster_data = data[cluster_candidates]
plt.scatter(cluster_data[:, 0], cluster_data[:, 1],
c=cluster_colors[i % len(cluster_colors)], alpha=0.7, s=30,
label=f'簇{cluster_idx}候选({len(cluster_candidates)}个)',
edgecolors='black', linewidths=0.5)
# 查询点
plt.scatter(query[0], query[1], c='#FFD93D', marker='*', s=200,
edgecolors='black', linewidth=2, label='查询点', zorder=10)
plt.title(f'4. 精细搜索:收集{len(candidate_indices)}个候选向量', fontweight='bold')
plt.legend(fontsize=7, loc='upper right')
plt.grid(True, alpha=0.3)
# 5. IVF结果
plt.subplot(2, 3, 5)
plt.scatter(data[:, 0], data[:, 1], c='lightgray', alpha=0.4, s=20)
plt.scatter(data[ivf_results, 0], data[ivf_results, 1],
c='red', marker='o', s=60, edgecolors='darkred',
linewidth=2, label='IVF结果')
for i, idx in enumerate(ivf_results[:5]):
plt.annotate(f'{i+1}', (data[idx, 0], data[idx, 1]),
xytext=(3, 3), textcoords='offset points',
fontsize=8, color='white', fontweight='bold')
plt.scatter(query[0], query[1], c='#FFD93D', marker='*', s=150,
edgecolors='black', linewidth=2, label='查询点')
plt.title('5. IVF结果', fontweight='bold')
plt.legend(fontsize=8)
plt.grid(True, alpha=0.3)
# 6. 结果对比
plt.subplot(2, 3, 6)
plt.scatter(data[:, 0], data[:, 1], c='lightgray', alpha=0.3, s=15)
plt.scatter(data[bf_results, 0], data[bf_results, 1],
c='green', marker='s', s=60, edgecolors='darkgreen',
linewidth=2, label='暴力搜索', alpha=0.7)
plt.scatter(data[ivf_results, 0], data[ivf_results, 1],
c='red', marker='o', s=50, alpha=0.8, label='IVF结果')
plt.scatter(query[0], query[1], c='#FFD93D', marker='*', s=150,
edgecolors='black', linewidth=2, label='查询点')
# 计算召回率
intersection = set(ivf_results) & set(bf_results)
recall = len(intersection) / len(bf_results)
plt.title(f'6. 结果对比 (召回率: {recall:.1%})', fontweight='bold')
plt.legend(fontsize=8)
plt.grid(True, alpha=0.3)
plt.tight_layout()
plt.show()
# 简化的统计信息
total_candidates = sum(len(ivf.inverted_lists[i]) for i in nearest_cluster_indices)
search_ratio = total_candidates / len(data)
print(f"
IVF算法统计:")
print(f"数据量: {len(data)} 个向量")
print(f"聚类数: {ivf.n_clusters} 个簇")
print(f"搜索簇: {ivf.n_probe} 个簇")
print(f"候选向量: {total_candidates} 个 ({search_ratio:.1%})")
print(f"召回率: {recall:.1%}")
return recall
#用少量样本进行可视化
ivf_tiny, data_tiny, query_tiny, ivf_results_tiny, bf_results_tiny = demonstrate_ivf(data_size=100)
recall = visualize_ivf(ivf_tiny, data_tiny, query_tiny, ivf_results_tiny, bf_results_tiny)结果输出: 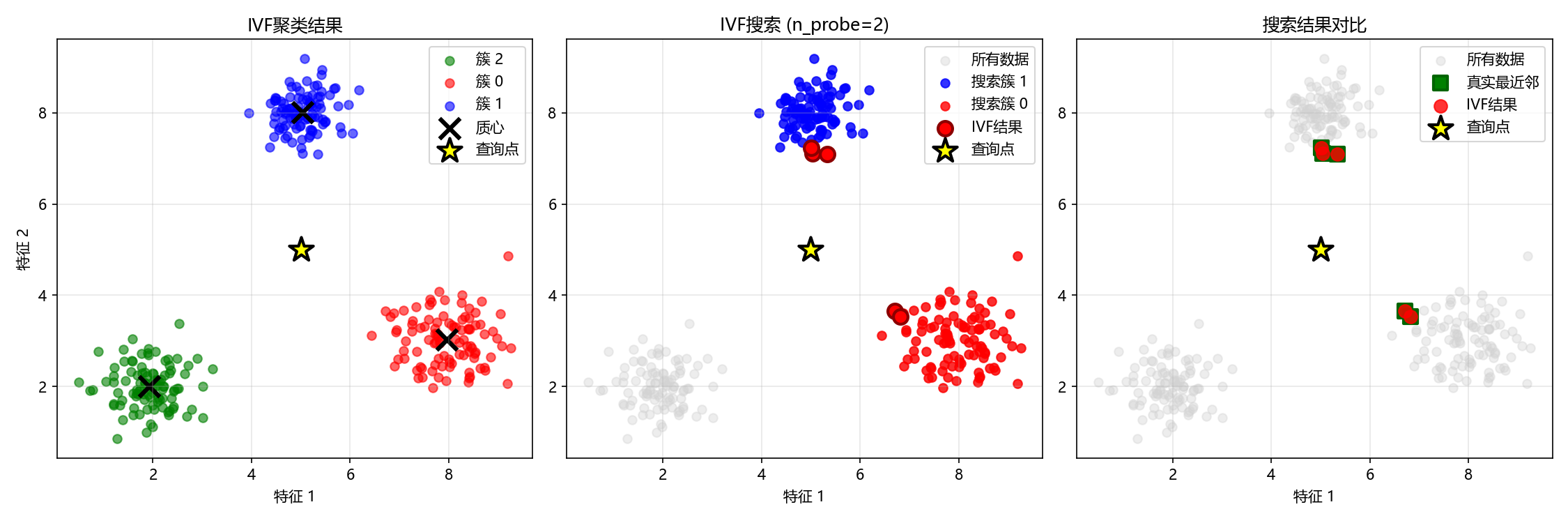
⚖️ 第六步:参数影响分析 让我们分析n_probe参数对搜索效果的影响:
def analyze_parameters():
"""分析n_probe参数对搜索效果的影响"""
data = generate_sample_data(1000, 2)
query = np.array([5.0, 5.0])
n_probe_values = [1, 2, 3]
results = []
for n_probe in n_probe_values:
ivf = SimpleIVF(n_clusters=5, n_probe=n_probe)
ivf.data = data
ivf.build_index(data)
# IVF搜索
start_time = time.time()
ivf_indices, _ = ivf.search(query, k=5, data=data)
ivf_time = time.time() - start_time
# 暴力搜索作为基准
bf_indices, _ = ivf.brute_force_search(query, k=5, data=data)
# 计算召回率
intersection = set(ivf_indices) & set(bf_indices)
recall = len(intersection) / len(bf_indices)
results.append({
'n_probe': n_probe,
'recall': recall,
'time': ivf_time,
'candidates_searched': sum(len(ivf.inverted_lists[i])
for i in range(n_probe))
})
# 显示结果
print("\n" + "="*50)
print("n_probe参数影响分析")
print("="*50)
for result in results:
print(f"n_probe={result['n_probe']}: "
f"召回率={result['recall']:.1%}, "
f"耗时={result['time']:.6f}秒, "
f"搜索向量数={result['candidates_searched']}")
return results
# 运行参数分析
parameter_results = analyze_parameters()结果输出:
开始训练IVF索引...
训练完成,得到5个簇
倒排索引构建完成「展示前5个簇」:
簇4: 333个向量
簇1: 100个向量
簇2: 110个向量
簇3: 123个向量
簇0: 334个向量
开始训练IVF索引...
训练完成,得到5个簇
倒排索引构建完成「展示前5个簇」:
簇0: 170个向量
簇1: 163个向量
簇3: 333个向量
簇4: 150个向量
簇2: 184个向量
开始训练IVF索引...
训练完成,得到5个簇
倒排索引构建完成「展示前5个簇」:
簇0: 333个向量
簇2: 153个向量
簇1: 180个向量
簇4: 191个向量
簇3: 143个向量
==================================================
n_probe参数影响分析
==================================================
n_probe=1: 召回率=80.0%, 耗时=0.001708秒, 搜索向量数=334
n_probe=2: 召回率=80.0%, 耗时=0.000682秒, 搜索向量数=333
n_probe=3: 召回率=100.0%, 耗时=0.000797秒, 搜索向量数=666从结论我们可以发现速度与精度的基本权衡关系 1.从 n_probe=2到 n_probe=3的变化,完美体现了IVF算法中速度与精度的经典权衡。
- 当 n_probe从2增加到3时,搜索的簇数量增加,因此需要计算的向量数量几乎翻倍(从333增加到666)。这导致搜索范围扩大,从而召回率从80%提升到了100%,但代价是耗时也有所增加(从0.000682秒增加到0.000797秒)。 这说明,增大 n_probe通常能以牺牲速度为代价来提升召回率。
2.发现异常点:n_probe=1的性能反常 这是一个非常关键的发现,这理论上,n_probe=1(只搜索1个最近的簇)应该是最快的,但结果却显示它最慢(0.001708秒)。
在 n_probe=1时,由于编程语言(如Python)的解释器开销、缓存未命中或其他偶然因素所致,你多尝试几次会发现n_probe=1的时间会有波动,这就是编译器本身编译或者缓存导致 最后,可以总结出以下核心要点:
基本规律成立:n_probe增大,搜索范围扩大,召回率提高,但耗时增加。
实践出真知:理论规律需要在实际测试中验证。实验中可能会出现像 n_probe=1这样的性能异常点,这正体现了参数调优和基准测试的重要性。
没有“最好”的参数,只有“最合适”的参数:最终的参数选择取决于应用场景对速度和精度的具体要求。