1.2 数据格式标准
"没有标准,就没有互操作性;没有互操作性,医学影像就只是孤岛上的数据。" —— 医学信息学的基石
想象这样一个场景:患者王先生在A医院做了CT检查,医生给了他一张光盘。几天后,他来到B医院寻求第二诊疗意见,却发现B医院的影像系统无法打开这张光盘。无奈之下,他只能在B医院重新拍片,不仅增加了费用,还承受了额外的辐射剂量。
这个看似荒谬的场景,在医学影像标准化之前却是常态。本章将带你了解医学影像数据格式标准的演进历史,重点介绍DICOM和NIfTI两大核心标准,以及它们如何解决了医学影像数据交换和共享的难题。
📜 标准化之前的混乱时代
各自为政的早期格式
在1970年代到1980年代,医学影像设备制造商各自开发了专有的数据格式。GE、Siemens、Philips、东芝等厂商都有自己的格式规范,彼此之间完全不兼容。
这种混乱带来了严重的问题:
- 医院层面:不同品牌设备的影像无法在同一系统中查看和管理
- 患者层面:转院就医时必须重新拍片,增加费用和辐射暴露
- 研究层面:多中心研究难以整合来自不同医院的影像数据
- 软件开发层面:第三方软件需要支持数十种格式,开发成本极高
| 时期 | 格式状况 | 主要问题 |
|---|---|---|
| 1970s-1980s | 每个厂商专有格式 | 完全不兼容,数据孤岛 |
| 1985-1988 | ACR-NEMA 1.0/2.0 | 仅支持点对点传输,缺乏网络能力 |
| 1993年 | DICOM 3.0发布 | 标准化时代开启 |
标准化的呼声与早期尝试
面对这种混乱局面,美国放射学会(ACR)和美国电气制造商协会(NEMA)在1983年开始合作,试图建立统一的医学影像标准。
早期标准的尝试:
- 1985年:ACR-NEMA 1.0发布,定义了基本的数据格式和点对点传输协议
- 1988年:ACR-NEMA 2.0发布,改进了数据字典和命令结构
为什么早期标准没有成功?
- 仅支持点对点的直接连接,无法适应网络化趋势
- 缺乏完善的信息模型,难以描述复杂的临床场景
- 没有明确的一致性测试标准,厂商实现各异
💡 标准化为何如此困难?
医学影像标准化不仅是技术问题,更是利益博弈的过程。设备厂商担心开放标准会削弱其市场竞争力,医院和放射科则迫切需要互操作性。DICOM的成功,在于找到了技术标准化与商业利益之间的平衡点。
🏥 DICOM标准——医学影像的通用语言
DICOM的诞生:从ACR-NEMA到DICOM 3.0
1993年,在吸取了ACR-NEMA标准的经验教训后,DICOM 3.0(Digital Imaging and Communications in Medicine)正式发布。这标志着医学影像标准化进入了新纪元。
DICOM 3.0的革命性突破:
- 网络传输支持:基于TCP/IP协议,适应互联网时代
- 面向对象的信息模型:清晰描述患者、检查、序列、图像的层次关系
- 标准化的服务类:定义了存储、查询、检索等标准操作
- 独立于厂商和设备:任何厂商都可以实现DICOM标准
🎯 DICOM的革命性意义
DICOM不仅仅是一个文件格式,而是一个完整的医学影像信息系统标准。它定义了数据格式、网络通信协议、服务类、一致性测试等方方面面,使得不同厂商的设备和系统能够无缝协作。
DICOM的核心架构
DICOM标准的核心是信息对象定义(IOD)和服务类(Service Classes)的组合。
信息对象定义(IOD)
IOD(Information Object Definition)定义了特定类型医学影像的数据结构。每种成像模态都有对应的IOD:
| IOD类型 | 描述 | 典型应用 |
|---|---|---|
| CT Image IOD | CT图像的数据结构 | CT扫描 |
| MR Image IOD | MRI图像的数据结构 | MRI扫描 |
| PET Image IOD | PET图像的数据结构 | PET扫描 |
| Ultrasound Image IOD | 超声图像的数据结构 | 超声检查 |
| RT Structure Set IOD | 放疗结构集 | 放疗计划 |
| Presentation State IOD | 显示状态 | 影像标注和测量 |
服务类(Service Classes)
服务类定义了DICOM系统之间的标准操作:
| 服务类 | 缩写 | 功能描述 |
|---|---|---|
| Storage Service Class | C-STORE | 存储影像到PACS |
| Query/Retrieve Service Class | C-FIND / C-MOVE | 查询和检索影像 |
| Worklist Service Class | MWL | 获取检查工作列表 |
| Print Service Class | C-PRINT | 打印影像到胶片 |
| Modality Performed Procedure Step | MPPS | 报告检查执行状态 |
服务对象对(SOP)
SOP(Service-Object Pair)是IOD和服务类的组合,代表一个具体的DICOM操作:
- SOP Class:定义了对象类型和可执行的操作
- SOP Instance:SOP Class的具体实例
- SOP Class UID:全球唯一标识符,如
1.2.840.10008.5.1.4.1.1.2(CT Image Storage)
示例: CT Image Storage SOP Class = CT Image IOD + Storage Service Class
DICOM文件结构深度解析
一个标准的DICOM文件由以下部分组成:
文件组成
┌─────────────────────────────────────┐
│ 文件前导码 (File Preamble) │ 128字节,通常为0x00
├─────────────────────────────────────┤
│ DICOM前缀 (DICOM Prefix) │ 4字节:"DICM"
├─────────────────────────────────────┤
│ 文件元信息 (File Meta Information) │ 传输语法、SOP Class UID等
├─────────────────────────────────────┤
│ 数据集 (Data Set) │ 实际的影像数据和元数据
│ ├─ 患者信息 │
│ ├─ 检查信息 │
│ ├─ 序列信息 │
│ ├─ 图像信息 │
│ └─ 像素数据 │
└─────────────────────────────────────┘数据元素(Data Element)
DICOM数据集由一系列数据元素(Data Element)组成,每个数据元素包含:
- 标签(Tag):(Group, Element)格式,如
(0010,0010)表示患者姓名 - 值表示(VR):Value Representation,数据类型
PN:Person Name(人名)DA:Date(日期,YYYYMMDD)TM:Time(时间,HHMMSS)UI:Unique Identifier(唯一标识符)US:Unsigned Short(无符号短整型)
- 值长度(VL):Value Length,数据长度
- 值字段(Value Field):实际数据
常用DICOM标签速查表
| 标签 | 名称 | VR | 描述 |
|---|---|---|---|
(0008,0018) | SOP Instance UID | UI | 图像唯一标识符 |
(0008,0060) | Modality | CS | 成像模态(CT/MR/PT等) |
(0010,0010) | Patient Name | PN | 患者姓名 |
(0010,0020) | Patient ID | LO | 患者ID |
(0010,0030) | Patient Birth Date | DA | 患者出生日期 |
(0010,0040) | Patient Sex | CS | 患者性别 |
(0020,000D) | Study Instance UID | UI | 检查唯一标识符 |
(0020,000E) | Series Instance UID | UI | 序列唯一标识符 |
(0020,0013) | Instance Number | IS | 图像编号 |
(0028,0010) | Rows | US | 图像行数 |
(0028,0011) | Columns | US | 图像列数 |
(0028,0030) | Pixel Spacing | DS | 像素间距(mm) |
(0028,0100) | Bits Allocated | US | 每像素分配位数 |
(7FE0,0010) | Pixel Data | OW/OB | 像素数据 |
⚠️ DICOM标签的命名规则
DICOM标签的Group号有特殊含义:
- 偶数Group(如0008, 0010):标准DICOM标签
- 奇数Group(如0009, 0011):厂商私有标签
- Group 0002:文件元信息
- Group 7FE0:像素数据
私有标签允许厂商扩展DICOM标准,但可能导致互操作性问题。
DICOM的层次结构:Patient-Study-Series-Instance
DICOM采用四层层次结构来组织医学影像数据:
Patient(患者)
└─ Study(检查)
└─ Series(序列)
└─ Instance(实例/图像)
各层次的含义:
| 层次 | 描述 | 关键UID | 示例 |
|---|---|---|---|
| Patient | 一个患者的所有检查 | Patient ID | 张三的所有影像 |
| Study | 一次就诊的所有影像 | Study Instance UID | 2024年1月15日的住院检查 |
| Series | 一次扫描的所有图像 | Series Instance UID | 胸部CT平扫序列 |
| Instance | 单张图像或单个对象 | SOP Instance UID | 第50层CT图像 |
实际应用示例:
- 患者"张三"(Patient)在2024年1月15日住院(Study)
- 进行了胸部CT检查,包含平扫(Series 1)和增强扫描(Series 2)
- 平扫序列包含300张图像(Instance 1-300)
📊 为什么需要层次结构?
层次结构使得医学影像数据的组织和管理更加清晰:
- 患者层面:追踪患者的完整影像历史
- 检查层面:关联同一次就诊的所有影像(CT、MRI、X射线等)
- 序列层面:区分不同的扫描协议(平扫、增强、不同序列)
- 实例层面:精确定位到每一张图像
这种结构也是PACS系统数据库设计的基础。
DICOM的传输语法与压缩
传输语法(Transfer Syntax)定义了DICOM数据的编码方式,包括字节序(大端/小端)和是否压缩。
常见传输语法
| 传输语法 | UID | 描述 | 应用场景 |
|---|---|---|---|
| Implicit VR Little Endian | 1.2.840.10008.1.2 | 隐式VR,小端字节序 | 默认传输语法 |
| Explicit VR Little Endian | 1.2.840.10008.1.2.1 | 显式VR,小端字节序 | 最常用 |
| Explicit VR Big Endian | 1.2.840.10008.1.2.2 | 显式VR,大端字节序 | 较少使用 |
| JPEG Baseline | 1.2.840.10008.1.2.4.50 | JPEG有损压缩 | 归档存储 |
| JPEG Lossless | 1.2.840.10008.1.2.4.70 | JPEG无损压缩 | 诊断影像 |
| JPEG 2000 Lossless | 1.2.840.10008.1.2.4.90 | JPEG 2000无损 | 高质量归档 |
| RLE Lossless | 1.2.840.10008.1.2.5 | 行程编码无损压缩 | 快速压缩 |
有损压缩 vs 无损压缩
📊 何时使用有损压缩?
无损压缩(推荐用于):
- 诊断影像:需要保留所有细节
- 放疗计划:精确的剂量计算
- 科研数据:定量分析
有损压缩(可用于):
- 归档存储:节省存储空间(压缩比可达10:1)
- 远程会诊:减少网络传输时间
- 教学影像:对细节要求不高
注意:许多国家和地区的医疗法规要求诊断影像必须使用无损压缩或不压缩。
🧠 NIfTI格式——神经影像的标准
NIfTI的诞生背景
虽然DICOM在临床应用中取得了巨大成功,但在科研领域,特别是神经影像学研究中,DICOM存在一些局限性:
DICOM在科研中的不便之处:
- 文件分散:一个3D体数据被分散在数百个DICOM文件中(每层一个文件)
- 元数据冗余:每个文件都包含完整的患者和检查信息
- 读写效率低:需要读取和解析大量文件
- 不适合批量处理:脚本处理复杂
神经影像学界的需求:
- 简单的文件结构(单文件存储3D/4D数据)
- 高效的读写性能
- 明确的空间坐标系统
- 适合脚本化批量处理
2004年,由美国国立卫生研究院(NIH)资助的神经影像信息学技术倡议(Neuroimaging Informatics Technology Initiative)发布了NIfTI-1格式,迅速成为神经影像研究的事实标准。
NIfTI文件结构
NIfTI-1格式
文件扩展名:
.nii:单文件格式(文件头+图像数据).hdr+.img:双文件格式(文件头和图像数据分离).nii.gz:压缩的单文件格式(推荐)
文件组成:
┌─────────────────────────────────────┐
│ NIfTI-1 Header │ 348字节,包含所有元数据
├─────────────────────────────────────┤
│ Extension (可选) │ 扩展信息
├─────────────────────────────────────┤
│ Image Data │ 图像数据(可多维)
└─────────────────────────────────────┘NIfTI文件头的关键字段
| 字段名 | 类型 | 描述 |
|---|---|---|
sizeof_hdr | int | 文件头大小(348字节) |
dim[8] | short | 图像维度(最多7维) |
datatype | short | 数据类型(uint8, int16, float32等) |
bitpix | short | 每像素位数 |
pixdim[8] | float | 体素尺寸(mm) |
vox_offset | float | 图像数据起始位置 |
scl_slope | float | 数据缩放斜率 |
scl_inter | float | 数据缩放截距 |
qform_code | short | 坐标系统方法1(四元数) |
sform_code | short | 坐标系统方法2(仿射矩阵) |
descrip | char[80] | 数据描述 |
NIfTI的坐标系统
NIfTI定义了明确的空间坐标系统,这是其相对于DICOM的重要优势之一。
两种坐标:
- 体素坐标(Voxel Coordinates):(i, j, k),整数索引
- 世界坐标(World Coordinates):(x, y, z),单位mm,相对于某个解剖参考点
坐标转换方法:
NIfTI提供两种方法将体素坐标转换为世界坐标:
Method 1(qform):基于四元数的刚体变换
- 适用于扫描仪坐标系
- 只包含旋转和平移
Method 2(sform):通用仿射变换矩阵
- 适用于标准空间(如MNI空间、Talairach空间)
- 可包含缩放、剪切等变换
仿射变换矩阵:
[x] [m11 m12 m13 m14] [i]
[y] = [m21 m22 m23 m24] [j]
[z] [m31 m32 m33 m34] [k]
[1] [ 0 0 0 1] [1]💡 为什么坐标系统如此重要?
在神经影像研究中,经常需要:
- 配准:将不同时间点或不同患者的影像对齐
- 标准化:将个体影像转换到标准空间(如MNI152)
- ROI分析:在标准空间中定义感兴趣区域
明确的坐标系统使得这些操作更加准确和可重复。
NIfTI-2:更大、更灵活
2011年,NIfTI-2格式发布,主要改进包括:
| 特性 | NIfTI-1 | NIfTI-2 |
|---|---|---|
| 文件头大小 | 348字节 | 540字节 |
| 维度数据类型 | int16 | int64 |
| 最大图像尺寸 | 32,767 | 9,223,372,036,854,775,807 |
| 体素尺寸精度 | float32 | float64 |
| 文件扩展名 | .nii | .nii (magic string区分) |
NIfTI-2主要用于超大规模影像数据(如全脑高分辨率连接组学数据)。
📁 其他常见医学影像格式
除了DICOM和NIfTI,医学影像领域还有一些其他格式,各有特定的应用场景。
格式对比总览
| 格式 | 文件扩展名 | 主要用途 | 优点 | 缺点 |
|---|---|---|---|---|
| DICOM | .dcm | 临床影像 | 标准化、元数据丰富、网络传输 | 文件分散、复杂 |
| NIfTI | .nii, .nii.gz | 神经影像研究 | 简单、高效、单文件 | 元数据有限 |
| NRRD | .nrrd, .nhdr | 3D Slicer | 灵活、支持多种数据类型 | 非标准化 |
| MINC | .mnc | MNI脑图谱 | 基于NetCDF/HDF5 | 使用范围有限 |
| Analyze 7.5 | .hdr, .img | 历史遗留 | 简单 | 左右方向歧义 |
| MetaImage | .mhd, .raw | ITK工具包 | 简单、灵活 | 非标准化 |
NRRD格式
全称:Nearly Raw Raster Data
特点:
- 简单的ASCII文件头 + 原始数据
- 支持多种数据类型和压缩方式
- 广泛用于3D Slicer等开源软件
文件结构:
.nhdr(文件头)+.raw(数据):双文件.nrrd:单文件
MINC格式
全称:Medical Image NetCDF
特点:
- 基于NetCDF(Network Common Data Form)
- MINC2基于HDF5
- 主要用于加拿大蒙特利尔神经研究所(MNI)
- 适合多中心研究和脑图谱构建
Analyze 7.5格式
历史地位:NIfTI的前身
问题:左右方向歧义(导致NIfTI的诞生)
现状:已被NIfTI取代,但仍有旧数据使用此格式
其他格式
- GIPL(Guys Image Processing Lab):早期医学影像格式
- PAR/REC:Philips MRI专有格式
- ECAT:PET专用格式
- MGH/MGZ:FreeSurfer软件格式
- BRIK/HEAD:AFNI软件格式
🔄 格式转换的必要性与注意事项
为什么需要格式转换?
医学影像处理的典型工作流程:
临床采集(DICOM) → 格式转换 → 研究分析(NIfTI等) → 结果可视化格式转换的常见场景:
- 临床到科研:DICOM → NIfTI(最常见)
- 软件兼容:不同软件工具要求不同格式
- 数据共享:标准化格式便于数据共享
- 存储优化:压缩格式节省存储空间
常用格式转换工具
| 工具 | 类型 | 主要功能 | 适用场景 |
|---|---|---|---|
| dcm2niix | 命令行 | DICOM → NIfTI | 批量转换,最流行 |
| pydicom | Python库 | DICOM读写 | 脚本化处理 |
| nibabel | Python库 | NIfTI/其他格式读写 | Python数据分析 |
| SimpleITK | Python/C++库 | 多格式转换 | 复杂处理流程 |
| 3D Slicer | 图形界面 | 多格式互转 | 交互式操作 |
| MRIcron | 图形界面 | DICOM → NIfTI | 简单易用 |
💡 工具选择建议
- 批量转换:dcm2niix(快速、准确)
- Python脚本:pydicom + nibabel
- 交互式操作:3D Slicer或MRIcron
- 复杂处理:SimpleITK
详细的工具使用方法将在下一节(1.3 常用开源工具)中介绍。
格式转换的注意事项
1. 元数据丢失问题
问题:DICOM包含丰富的临床元数据(患者信息、检查参数、设备信息等),而NIfTI只保留影像相关的基本信息。
解决方案:
- 保留原始DICOM文件作为备份
- 使用工具(如dcm2niix)导出JSON格式的元数据
- 在数据库中记录DICOM与NIfTI的对应关系
2. 坐标系统转换
问题:DICOM使用LPS坐标系(Left-Posterior-Superior),而NIfTI通常使用RAS坐标系(Right-Anterior-Superior)。
注意:
- 确保转换工具正确处理坐标系统转换
- 验证转换后的图像方向(左右、前后、上下)
- 检查仿射矩阵是否正确
3. 多序列处理
问题:一个DICOM Study可能包含多个Series(平扫、增强、不同序列等)。
策略:
- 按Series分别转换
- 保持清晰的文件命名规则
- 记录Series描述和序列号
4. 数据隐私与匿名化
问题:DICOM文件包含患者隐私信息(姓名、ID、出生日期等)。
匿名化要求:
- 去除或替换患者姓名、ID
- 移除出生日期或转换为年龄
- 保留必要的临床信息(性别、检查日期等)
- 遵守HIPAA、GDPR等隐私法规
⚠️ 数据隐私与匿名化
在共享医学影像数据前,必须进行匿名化处理:
- 必须移除:患者姓名、ID、地址、电话
- 可选移除:出生日期(可转换为年龄)、检查日期(可转换为相对日期)
- 应保留:性别、年龄、检查类型、影像参数
许多国家和地区对医学数据共享有严格的法律要求,违规可能导致严重后果。
🌐 DICOM网络传输与PACS系统
DICOM网络协议
DICOM不仅定义了文件格式,还定义了网络传输协议,使得医学影像设备和系统能够通过网络进行通信。
核心概念
Application Entity (AE):DICOM网络节点
- AE Title:节点标识符(如"CT_SCANNER_1")
- IP地址和端口:网络地址(默认端口104)
Association:两个AE之间的连接
- 类似于TCP连接,但在应用层
- 需要协商传输语法和SOP Class
DICOM网络服务
| 服务命令 | 功能 | 典型应用 |
|---|---|---|
| C-ECHO | 测试连接 | DICOM Ping,验证网络连通性 |
| C-STORE | 存储影像 | 影像设备发送影像到PACS |
| C-FIND | 查询影像 | 工作站查询PACS中的影像 |
| C-MOVE | 检索影像 | 工作站从PACS检索影像 |
| C-GET | 获取影像 | 类似C-MOVE,但直接返回影像 |
PACS系统简介
PACS(Picture Archiving and Communication System)是医院影像归档与通信系统,是现代医院数字化影像管理的核心。
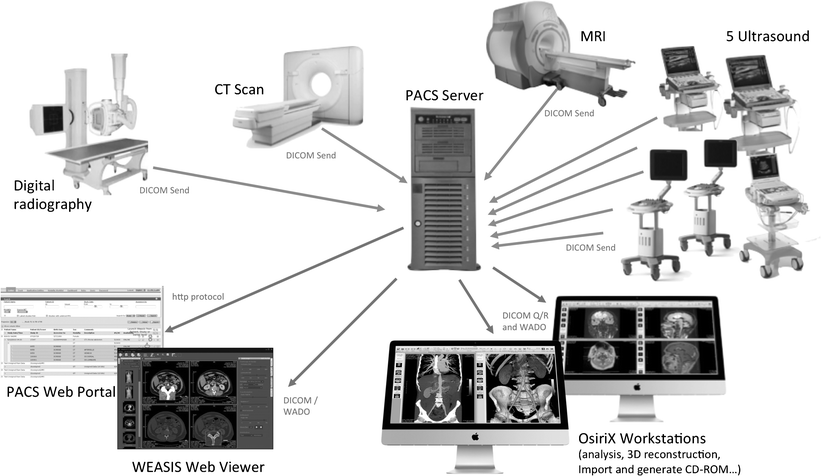 典型PACS系统的架构组成
典型PACS系统的架构组成
PACS的组成
┌─────────────────────────────────────────────────────┐
│ PACS系统 │
├─────────────────────────────────────────────────────┤
│ 影像采集设备 │
│ ├─ CT扫描仪 │
│ ├─ MRI扫描仪 │
│ ├─ X射线设备 │
│ └─ 超声设备 │
├─────────────────────────────────────────────────────┤
│ PACS服务器 │
│ ├─ 数据库服务器(元数据) │
│ ├─ 存储服务器(影像数据) │
│ └─ Web服务器(远程访问) │
├─────────────────────────────────────────────────────┤
│ 诊断工作站 │
│ ├─ 放射科医生工作站 │
│ ├─ 临床医生查看终端 │
│ └─ 移动设备(平板、手机) │
└─────────────────────────────────────────────────────┘PACS工作流程
- 影像采集:CT/MRI等设备完成扫描
- 影像传输:设备通过DICOM C-STORE发送影像到PACS
- 影像存储:PACS服务器存储影像并建立索引
- 影像查询:医生通过工作站查询患者影像(C-FIND)
- 影像检索:工作站从PACS检索影像(C-MOVE)
- 影像诊断:医生查看影像并出具报告
- 影像归档:长期存储和备份
🏥 现代医院的数字化影像流程
PACS系统彻底改变了医院的影像管理方式:
- 无胶片化:从传统胶片到数字影像
- 即时访问:医生可在任何终端查看影像
- 远程会诊:专家可远程查看和诊断
- 长期归档:数字化存储,永不丢失
- AI辅助:为AI辅助诊断提供数据基础
一个大型三甲医院的PACS系统每天可能处理数千次检查,存储数TB的影像数据。
🚀 未来趋势与新兴标准
DICOMweb:Web时代的DICOM
传统DICOM网络协议基于TCP/IP的自定义协议,在现代Web环境中存在一些局限性。DICOMweb是基于RESTful API的新一代DICOM服务标准。
DICOMweb的三大服务:
- WADO-RS(Web Access to DICOM Objects):通过HTTP检索DICOM对象
- QIDO-RS(Query based on ID for DICOM Objects):通过HTTP查询DICOM对象
- STOW-RS(Store Over the Web):通过HTTP存储DICOM对象
优势:
- 跨平台、跨语言
- 易于集成到Web应用
- 云端友好
- 支持现代Web技术(JSON、OAuth等)
FHIR与医学影像
FHIR(Fast Healthcare Interoperability Resources)是新一代医疗信息交换标准,由HL7组织开发。
FHIR ImagingStudy资源:
- 描述医学影像检查的元数据
- 与DICOM互操作
- 支持RESTful API
DICOM与FHIR的互补:
- DICOM:专注于影像数据和影像特定的工作流程
- FHIR:覆盖更广泛的医疗信息(病历、检验、处方等)
- 两者结合:实现完整的医疗信息互操作性
云端医学影像与AI
未来趋势:
- 云PACS:影像数据存储在云端,随时随地访问
- AI辅助诊断:深度学习模型直接集成到PACS工作流程
- 标准化数据集:公开的标准化影像数据集(如TCIA、UK Biobank)
- 联邦学习:在保护隐私的前提下进行多中心AI模型训练
💡 关键要点总结
标准化的必要性:早期医学影像格式混乱,DICOM标准的诞生解决了互操作性问题,使得不同厂商的设备和系统能够无缝协作。
DICOM的核心架构:IOD(信息对象定义)+ Service Class(服务类)= SOP(服务对象对),这是DICOM标准的基础。
DICOM文件结构:文件前导码 + "DICM"前缀 + 文件元信息 + 数据集(由标签-值对组成)。
DICOM层次结构:Patient-Study-Series-Instance四层结构,清晰组织医学影像数据。
NIfTI的优势:简单、高效、单文件存储3D/4D数据,明确的空间坐标系统,成为神经影像研究的标准格式。
格式转换的重要性:临床数据(DICOM)与研究数据(NIfTI)之间的转换是医学影像处理的常见需求。
格式转换注意事项:元数据丢失、坐标系统转换、多序列处理、数据隐私匿名化。
DICOM网络与PACS:DICOM不仅是文件格式,更是完整的网络通信协议,PACS系统是现代医院数字化影像管理的核心。
未来趋势:DICOMweb、FHIR、云PACS、AI辅助诊断,医学影像标准化和智能化的发展方向。
💡 下一步学习
现在你已经了解了医学影像的数据格式标准。在下一节(1.3 常用开源工具)中,我们将深入探讨如何使用Python、ITK、SimpleITK、3D Slicer等工具进行医学影像处理。在第2章中,我们将学习医学影像的预处理技术,包括去噪、配准、分割等实用方法。