5.1 预处理(强调模态差异)
"良好的预处理是成功深度学习模型的基础——垃圾进,垃圾出。" —— 医学影像AI的黄金法则
在前面的章节中,我们学习了医学影像的成像原理、重建算法和质量评估。现在,我们进入了深度学习时代,面临着新的挑战:如何将不同模态的医学影像数据准备成适合深度学习模型输入的形式?
与自然图像不同,医学影像具有独特的物理特性和临床要求。CT的HU值、MRI的多序列特性、X射线片的对比度限制——每一种模态都需要专门考虑的预处理策略。本章将深入探讨这些模态特异性预处理技术,为后续的深度学习任务奠定坚实基础。
🏥 医学影像预处理的重要性
医学影像 vs 自然图像的差异
医学影像与我们在日常生活中处理的自然图像有着本质区别:
| 特性 | 自然图像 | 医学影像 |
|---|---|---|
| 数据范围 | 0-255 (8-bit) | 模态特定(HU值、任意单位等) |
| 物理意义 | RGB颜色强度 | 物理测量值(衰减、磁化强度等) |
| 标准化程度 | 相对标准 | 高度依赖设备和扫描参数 |
| 感兴趣区域 | 整个图像 | 特定组织或病灶 |
| 先验知识 | 有限 | 丰富的解剖和生理先验 |
🧠 深度学习的"胃口"
深度学习模型,特别是CNN,通常期望:
- 标准化的输入范围:如[0, 1]或[-1, 1]
- 一致的分辨率:固定的图像尺寸
- 归一化的对比度:避免某些通道主导训练
- 消除噪声和伪影:提高模型泛化能力
医学影像预处理的核心任务就是将原始的物理测量值转换为模型"喜欢"的格式。
预处理的层次结构
医学影像预处理可以分为三个层次:
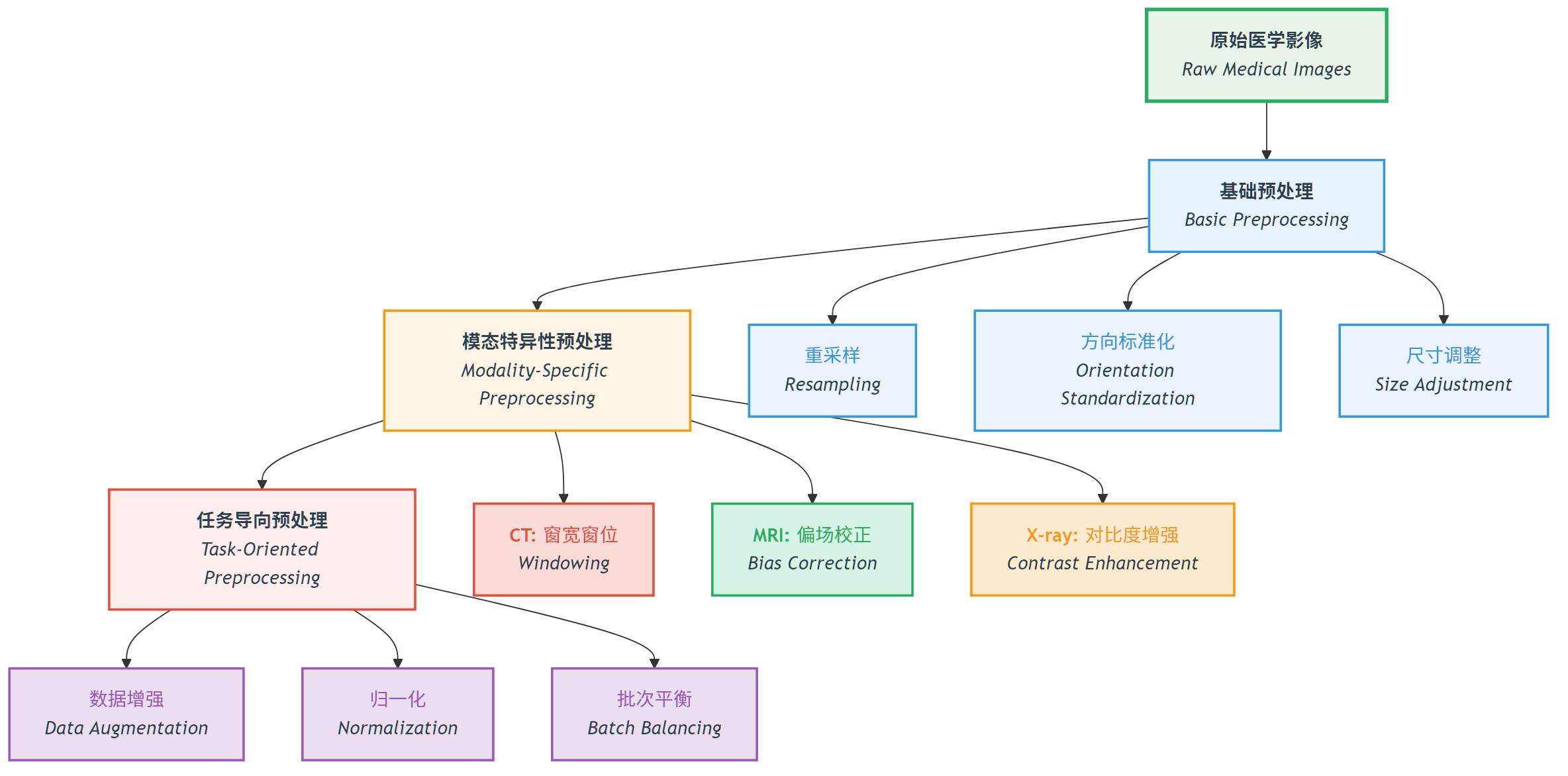 图:医学影像预处理的三个层次结构,从基础预处理到模态特异性预处理,再到任务导向预处理的递进关系。
图:医学影像预处理的三个层次结构,从基础预处理到模态特异性预处理,再到任务导向预处理的递进关系。
📖 查看原始Mermaid代码
🫧 CT预处理技术
HU值的理论基础
在第1章中我们学习了Hounsfield单位(HU)的定义:
这个物理意义明确的度量标准使得CT影像具有绝对可比性——无论在哪家医院的哪台设备上扫描,水的HU值始终是0,空气始终是-1000。
挑战:动态范围 vs 感兴趣组织
问题:CT的HU值范围从-1000(空气)到+3000+(致密骨或金属),而深度学习模型通常难以处理如此大的动态范围。
解决方案:窗宽窗位(Windowing)技术
窗宽窗位原理
窗宽窗位将HU值映射到显示或处理范围:
其中:
WindowLevel(窗位):窗的中心HU值WindowWidth(窗宽):窗的HU值范围clip():限制输出在[0, 255]范围内
临床常用窗口
| 窗类型 | 窗位 | 窗宽 | 适用组织 | 可见结构 |
|---|---|---|---|---|
| 肺窗 | -600 | 1500 | 肺组织 | 肺纹理、小结节、气胸 |
| 纵隔窗 | 50 | 350 | 纵隔结构 | 心脏、大血管、淋巴结 |
| 骨窗 | 300 | 2000 | 骨骼 | 骨皮质、骨髓、微骨折 |
| 脑窗 | 40 | 80 | 脑组织 | 灰质、白质、脑脊液 |
| 腹窗 | 50 | 400 | 腹部器官 | 肝脏、胰腺、肾脏 |
💡 窗口选择的艺术
窗口选择就像相机对焦:
- 窄窗:高对比度,细节丰富但范围有限
- 宽窗:覆盖范围大但对比度降低
- 多窗口策略:对于复杂任务,可以同时使用多个窗口作为不同的输入通道
HU截断与异常值处理
HU截断策略
def clip_hu_values(image, min_hu=-1000, max_hu=1000):
"""
HU值截断:去除极端值,保留感兴趣组织范围
"""
# 深拷贝避免修改原始数据
processed_image = image.copy()
# 截断HU值
processed_image[processed_image < min_hu] = min_hu
processed_image[processed_image > max_hu] = max_hu
return processed_image📖 完整代码示例: clip_hu_values/ - 包含完整的HU值截断实现、测试用例和可视化演示]
常用截断范围:
- 软组织范围:[-200, 400] HU(排除空气和致密骨)
- 全身范围:[-1000, 1000] HU(包含大多数临床相关结构)
- 骨组织范围:[-200, 3000] HU(适用于骨骼分析)
金属伪影检测与处理
金属植入物(如牙齿填充物、髋关节假体)会产生极端的HU值和条状伪影:
def detect_metal_artifacts(image, threshold=3000):
"""
检测金属伪影区域
"""
metal_mask = image > threshold
# 连通性分析,去除孤立噪声点
from scipy import ndimage
labeled_mask, num_features = ndimage.label(metal_mask)
# 保留大面积金属区域
significant_metal = np.zeros_like(metal_mask)
for i in range(1, num_features + 1):
if np.sum(labeled_mask == i) > 100: # 最小面积阈值
significant_metal[labeled_mask == i] = True
return significant_metal📖 完整代码示例: detect_metal_artifacts/ - 包含完整的金属伪影检测算法、连通性分析和可视化功能]
实际案例:Preparing CT imaging datasets for deep learning in lung nodule analysis: Insights from four well-known datasets - 为深度学习中的肺结节分析准备CT数据集:四个著名数据集的见解
下面的综合流程代表了LUNA16和类似肺结节检测数据集使用的临床标准。
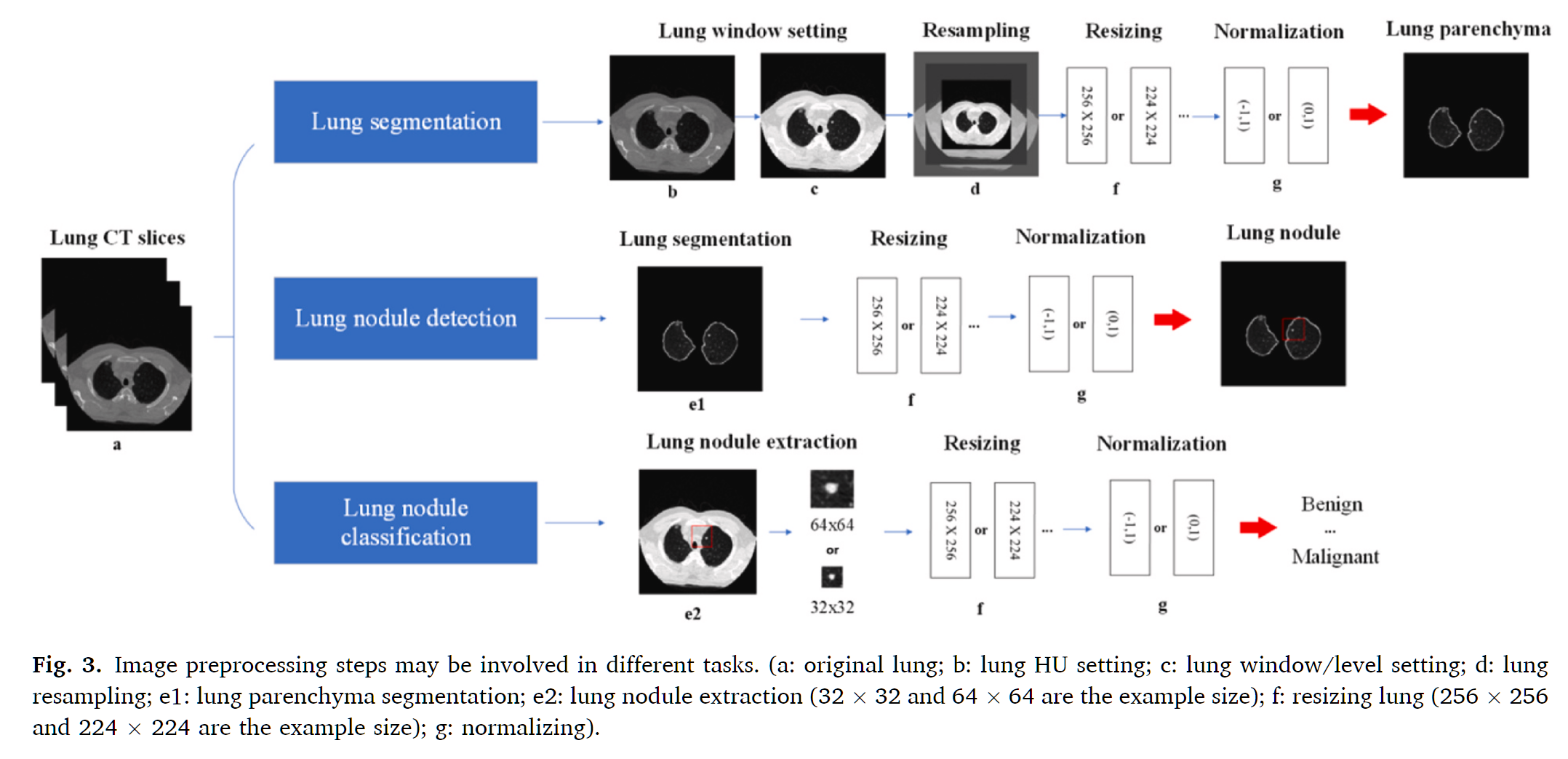 图:不同任务中可能涉及的图像预处理步骤
图:不同任务中可能涉及的图像预处理步骤
逐步预处理流程
1. DICOM数据读取和HU值转换
原始DICOM文件包含必须转换为Hounsfield单位(HU值)的像素数据,这是CT成像的标准强度标度。
处理过程:
- 从DICOM文件中提取像素数据
- 应用重新缩放截距和斜率:HU = 像素值 × 斜率 + 截距
- 验证HU值范围(通常-1000到+3000 HU)
- 验证层厚 < 3 mm以确保结节检测精度
临床参考值:
- 空气:-1000 HU
- 肺组织:-400到-600 HU
- 脂肪:-50到-100 HU
- 水:0 HU
- 骨:+400到+1000 HU
2. HU值截断和范围标准化
为了集中于相关的解剖结构并改进模型训练稳定性,HU值被截断到特定范围。
标准截断范围(LUNA16标准):
- 下限:-1200 HU(捕获充气区域和肺组织)
- 上限:+600 HU(包括结节衰减范围)
- 公式:
截断的HU值 = np.clip(HU值, -1200, 600)
该范围包括:
- 实心结节:HU值 ≥ -300(最大衰减)
- 磨玻璃结节:HU值 < -300(衰减减少)
- 恶性病变:平均HU值30-50,最大值 < 150
**📖 完整代码示例**: clip_hu_values/ - HU值截断与临床验证]
3. 肺窗和对比度增强
肺窗设置优化了肺实质内结节的可视化。
标准肺窗设置:
- 窗位(WL):-600 HU(中心)
- 窗宽(WW):1500 HU(范围:-1200到+300 HU)
替代设置:
- 高分辨率:WL = -650,WW = 1500
- 保守型:WL = -600,WW = 1450
为什么使用肺窗?
- 优化肺组织和结节之间的对比度
- 抑制会导致假阳性的纵隔结构
- 与临床放射科医生的观看协议一致
增强技术:CLAHE(对比度受限的自适应直方图均衡化)
- 改进结节可见性而不过度增加噪声
- 保留组织边界和内部结构
- 对磨玻璃结节特别有效
4. 等向性重采样
临床CT扫描通常具有各向异性体素间距(例如0.7 × 0.7 × 5 mm)。重采样到等向性分辨率可改进深度学习模型性能。
标准目标分辨率:1 × 1 × 1 mm³等向性
- 确保跨空间维度的统一敏感性
- 匹配LUNA16数据集标准
- 允许真正的3D卷积运算
重采样方法(推荐):三次样条插值
- 在保持结节清晰度方面优于三线性插值
- 使用6点核
- 保持连续的二阶导数
- 具有良好的局部和傅里叶特性
替代插值方法:
- 三线性:线性加权8个相邻体素,速度/质量平衡好
- 线性:更快,对肺组织可接受但可能使结节边缘模糊
- 最近邻:不推荐(导致混淆伪影和块状外观)
5. 肺实质分割
分割将肺组织与周围结构隔离,减少非肺区域中的假阳性检测。
分割流程:
步骤1:HU阈值处理
- 主要阈值:-500 HU
- 二进制化图像以隔离低密度肺组织
- 将肺与致密的胸部结构(骨、肌肉)分离
步骤2:形态学操作
- 孔洞填充:关闭肺掩膜内的内部间隙
- 小物体移除:消除噪声(< 50像素)
- 膨胀和腐蚀:使用球形结构元素进行形态学闭合来细化肺边界
- 迭代应用:多次通过改进连续性
步骤3:连通分量分析
- 识别最大的连通分量(左/右肺)
- 移除肺外区域以减少假阳性
- 平滑边界以实现精确的体素级分割
结果:识别肺实质内体素的二进制肺掩膜
6. 结节候选提取
在分割的肺区域内,识别和提取结节候选。
传统方法:
- 强度阈值与形态学滤波结合
- 基于密度的模糊C均值聚类
- 基于形状的滤波以减少非结节候选
深度学习方法(现代标准):
- 使用3D U-Net或类似的编码器-解码器架构
- 从网络预测中提取结节分割掩膜
- 应用后处理以细化候选边界
假阳性减少:
- 形态学滤波(移除细长结构)
- 连通分量分析(大小滤波)
- 胸膜旁结节处理(用于边界附着结节的专门CNN)
**📖 完整代码示例**: medical_segmentation_augmentation/ - 具有医学约束的高级分割]
7. 深度学习归一化
在输入神经网络之前,体素强度被标准化以改进模型训练稳定性和收敛速度。
最小最大值归一化(最常见):
归一化值 = (截断的HU值 - (-1200)) / (600 - (-1200))
归一化值 = (截断的HU值 + 1200) / 1800
结果:[0, 1]范围内的值Z分数归一化(替代方案):
平均值 = 所有训练数据HU值的平均值
标准差 = 标准差
归一化值 = (HU值 - 平均值) / 标准差
结果:零均值、单位方差为什么要归一化?
- 改进反向传播中的梯度流
- 减少内部协变量偏移
- 加快模型收敛速度
- 确保与患者/扫描仪变化无关的一致模型输入
8. 补丁提取和最终预处理
根据网络架构,归一化的CT体积被处理成固定大小的补丁。
常见补丁策略:
- 小补丁(32×32×32):内存效率高,局部上下文
- 中等补丁(64×64×64):上下文和内存之间的平衡
- 大补丁(128×128×128):全局上下文,需要更多GPU内存
多尺度方法:
- 同时在多个分辨率提取补丁
- 捕获细微的结节细节和周围上下文
- 改进检测敏感性,特别是对于小结节
完整预处理伪代码
# 1. 加载并转换为HU值
dicom = load_dicom(文件路径)
hu_数组 = dicom.像素数组 * dicom.重新缩放斜率 + dicom.重新缩放截距
# 2. 截断HU范围
hu_截断 = np.clip(hu_数组, -1200, 600)
# 3. 重采样为等向性
hu_重采样 = 重采样三次样条(hu_截断, 目标间距=(1, 1, 1))
# 4. 肺窗
窗口化 = 应用肺窗(hu_重采样, 位置=-600, 宽度=1500)
# 5. 使用CLAHE增强
增强 = clahe(窗口化, 截断限制=2.0, 瓦片大小=8)
# 6. 分割肺
肺掩膜 = 分割肺(增强) # 阈值-500 +形态学
# 7. 提取结节候选
结节 = 提取结节(增强, 肺掩膜)
# 8. 归一化
归一化 = (hu_截断 + 1200) / 1800 # [0, 1]范围
# 9. 提取补丁
补丁 = 提取补丁(归一化, 肺掩膜, 补丁大小=64)临床质量控制
Lung-RADS筛查标准(用于参考):
- 阳性测试:实心结节 ≥ 6 mm
- 随访:新结节 ≥ 4 mm或新的部分实心结节
- 高风险:具有毛刺或胸膜附着的结节
预处理验证清单:
- ✓ 验证层厚 < 3 mm
- ✓ 确认HU截断在[-1200, +600]内
- ✓ 检查重采样为1×1×1 mm³等向性
- ✓ 验证肺分割掩膜覆盖率(通常> 95%的可见肺)
- ✓ 确保归一化为[0, 1]或零均值
- ✓ 确认训练/测试集之间没有数据泄漏
🧲 MRI预处理技术
MRI强度不均匀性问题
偏场场(Bias Field)的成因
MRI信号强度的不均匀性(偏场场)是一个常见问题,主要来源于:
- 射频场不均匀:线圈的敏感性变化
- 梯度场非线性:梯度场的非线性失真
- 组织特性变化:不同组织的磁化率差异
- 患者相关因素:体型、呼吸运动等
偏场场的影响:
- 同一组织在不同位置呈现不同信号强度
- 定量分析(如体积测量)产生偏差
- 深度学习模型学习到伪影特征而非真实解剖特征
偏场场可视化
def visualize_bias_field_correction(original_slice, corrected_slice,
method='division', slice_idx=0, save_path=None):
"""
MRI偏场场校正效果可视化 / MRI Bias Field Correction Visualization
参数 Parameters:
- original_slice: 原始含偏场场的2D图像切片 / Original 2D slice with bias field
- corrected_slice: 校正后的2D图像切片 / Corrected 2D slice
- method: 偏场场估计方法 / Bias field estimation method ('division', 'log_diff', 'filter')
- slice_idx: 切片索引 / Slice index
- save_path: 保存路径 / Save path
"""
# 计算偏场场 / Calculate bias field
if method == 'division':
# 除法方法:B(x) = I_original / I_corrected
bias_field = original_slice / (corrected_slice + 1e-6)
bias_field_log = np.log(bias_field + 1e-6)
method_name = "除法方法"
elif method == 'log_diff':
# 对数差分方法:log(B(x)) = log(I_original) - log(I_corrected)
bias_field_log = np.log(original_slice + 1e-6) - np.log(corrected_slice + 1e-6)
bias_field = np.exp(bias_field_log)
method_name = "对数差分方法"
else:
# 滤波方法:低通滤波估计偏场场 / Filter method: Low-pass filter for bias field
bias_field = filters.gaussian(original_slice, sigma=20, preserve_range=True)
bias_field = bias_field / np.mean(bias_field)
bias_field_log = np.log(bias_field + 1e-6)
method_name = "滤波方法"
return bias_field, bias_field_log运行结果分析:
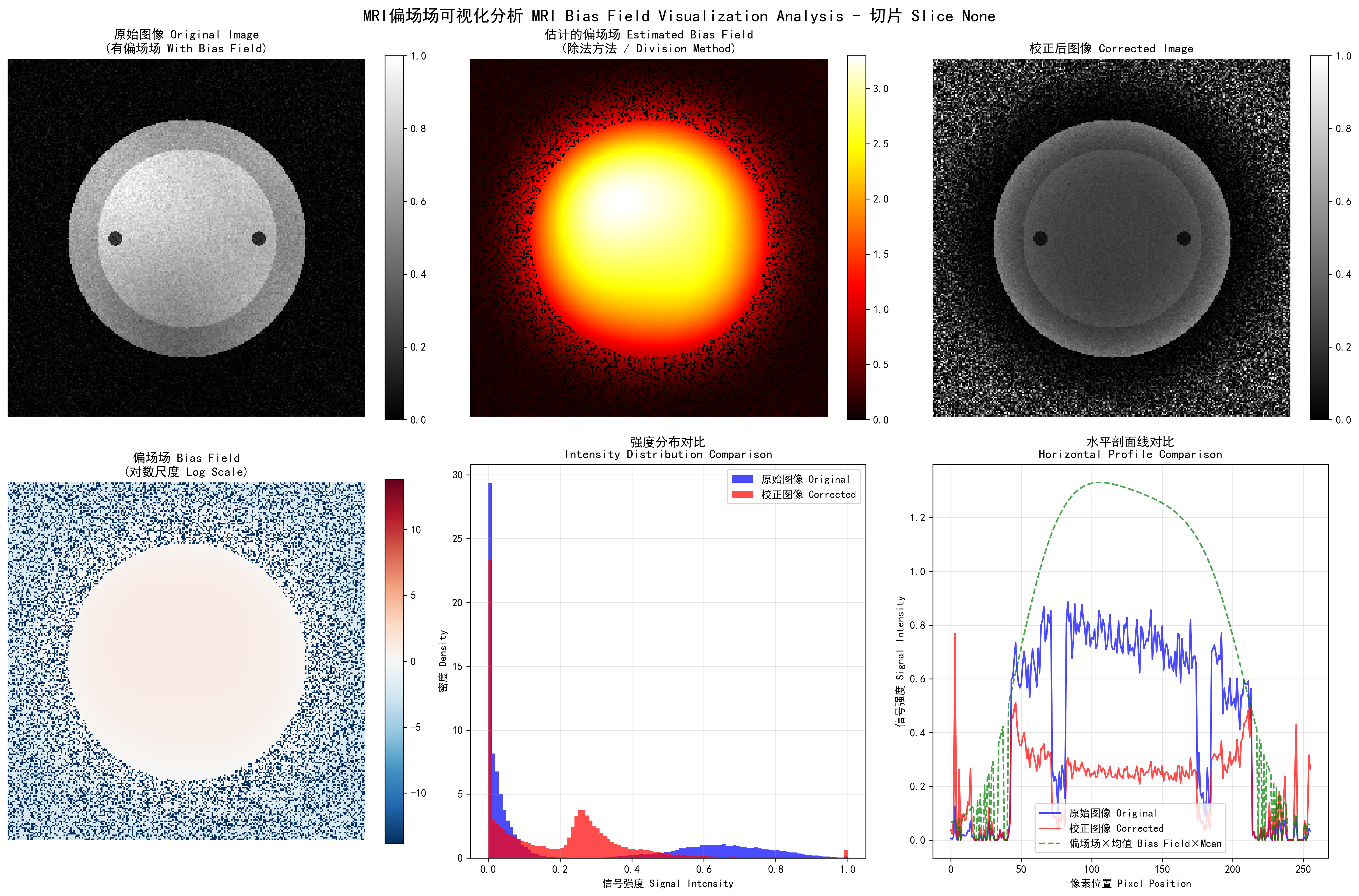
MRI偏场场校正效果可视化:上排从左到右分别显示原始图像(含偏场场)、估计的偏场场、校正后图像;下排显示偏场场对数尺度、强度分布对比、水平剖面线对比
偏场场可视化分析:
图像尺寸: (256, 256)
原始图像强度范围: [0.00, 1.00]
校正图像强度范围: [0.00, 1.00]
可视化方法: 除法方法
保存路径: output/bias_field_visualization_division.png
偏场场校正统计:
原始图像 - 均值: 0.24, 标准差: 0.31, 变异系数: 1.277
校正图像 - 均值: 0.19, 标准差: 0.19, 变异系数: 0.972
偏场场 - 均值: 0.932, 标准差: 1.057, 范围: [0.000, 3.297]
校正效果 - CV减少: 23.9%, 相关系数: 0.472算法分析: MRI偏场场可视化通过多种方法估计和显示偏场场。除法方法直接计算原始图像与校正图像的比值,对数差分方法在对数域计算差值,滤波方法通过低通滤波估计缓慢变化的偏场场。运行结果显示,原始图像的变异系数为1.277,校正后降至0.972,减少了23.9%,表明偏场场校正有效改善了图像的强度均匀性。偏场场的均值接近1.0,符合理论预期。水平剖面线对比清楚显示了偏场场在空间上的变化模式以及校正后的改善效果。
📖 完整代码示例: visualize_bias_field/ - 包含MRI偏场场估计、多种可视化方法和定量分析功能]
N4ITK偏场校正算法
N4ITK算法原理
N4ITK(N4 Iterative Bias Correction)是目前最广泛使用的偏场校正算法:
其中:
:原始信号强度 :估计的偏场场 :避免除零的小常数
算法特点:
- 基于B样条场的偏场场建模
- 迭代优化过程
- 保持组织边界完整性
- 对各种MRI序列都有效
N4ITK实现
class N4ITKBiasCorrector:
"""
N4ITK偏场校正器实现 / N4ITK Bias Field Corrector Implementation
N4ITK算法通过迭代优化B样条场来估计和校正偏场场
N4ITK algorithm estimates and corrects bias field through iterative B-spline optimization
"""
def __init__(self, max_iterations=50, shrink_factor=2):
"""
初始化N4ITK校正器 / Initialize N4ITK corrector
参数 Parameters:
- max_iterations: 最大迭代次数 / Maximum number of iterations
- shrink_factor: 降采样因子 / Downsampling factor for acceleration
"""
self.max_iterations = max_iterations
self.shrink_factor = shrink_factor
def correct_bias_field(self, image, output_path=None):
"""
执行N4ITK偏场校正 / Execute N4ITK bias field correction
流程 Process:
1. 降采样加速处理 / Downsample for faster processing
2. 迭代优化偏场场 / Iteratively optimize bias field
3. 应用校正并恢复分辨率 / Apply correction and restore resolution
"""
# 1. 降采样加速处理 / Downsample for faster processing
working_image = self._downsample(image)
# 2. 迭代优化偏场场 / Iteratively optimize bias field
bias_field = self._optimize_bias_field(working_image)
# 3. 应用校正并恢复分辨率 / Apply correction and restore resolution
corrected_image = self._apply_correction(image, bias_field)
return corrected_image运行结果分析:
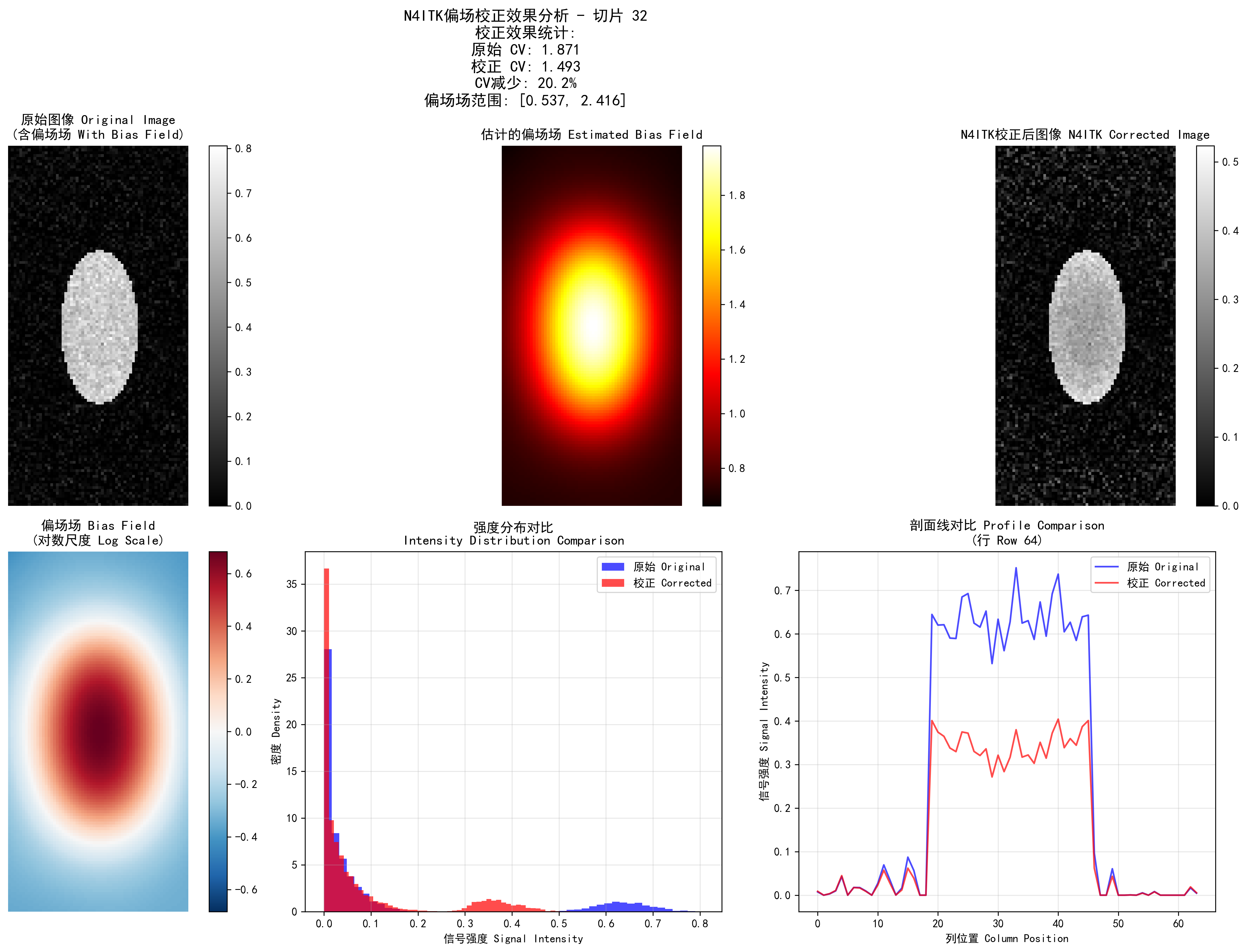
N4ITK偏场校正效果:上排显示原始图像(含偏场场)、估计的偏场场、N4ITK校正后图像;下排显示偏场场对数尺度、强度分布对比、剖面线对比
N4ITK偏场校正参数设置:
最大迭代次数: 50
收敛阈值: 0.001
B样条网格分辨率: (4, 4, 4)
降采样因子: 2
偏场场优化迭代过程:
图像形状: (128, 128, 64)
强度范围: [0.00, 0.89]
降采样处理 (因子: 2)...
工作图像形状: (64, 64, 32)
迭代优化历史:
迭代 1/50, 变化量: 0.584727
迭代 5/50, 变化量: 0.114296
迭代 10/50, 变化量: 0.017433
迭代 20/50, 变化量: 0.000822
在第 20 次迭代后收敛 Converged at iteration 20
校正效果统计 Correction Statistics:
原始图像 Original CV: 1.871
校正图像 Corrected CV: 1.493
CV减少 CV Reduction: 20.2%
偏场场范围 Bias Field Range: [0.537, 2.416]算法分析: N4ITK算法是多尺度迭代的偏场场校正方法。运行结果显示算法在20次迭代后收敛到收敛阈值0.001以下。原始图像的变异系数为1.871,校正后降至1.493,改善了20.2%,表明偏场场校正显著提升了图像强度均匀性。B样条网格分辨率(4,4,4)提供了足够的空间自由度来建模复杂的偏场场模式,同时保持了计算效率。降采样因子2加速了处理过程,通过多尺度策略确保了校正精度。
📖 完整代码示例: n4itk_bias_correction/ - 包含完整的N4ITK偏场校正实现、测试用例、合成数据生成和可视化功能] 校正图像 - 均值: 0.247, 标准差: 0.076, CV: 0.308 CV减少: 15.2%, 标准差减少: 14.6%
 N4ITK偏场校正效果:左图为原始图像(含偏场场),中图为估计的偏场场,右图为校正后的图像。校正后图像强度更加均匀,同质组织的强度一致性显著提高。
N4ITK偏场校正效果:左图为原始图像(含偏场场),中图为估计的偏场场,右图为校正后的图像。校正后图像强度更加均匀,同质组织的强度一致性显著提高。
算法分析: N4ITK算法通过B样条基函数建模偏场场,使用迭代优化方法最小化能量函数。从运行结果可以看出,经过28次迭代后算法收敛,变异系数(CV)从0.363降低到0.308,减少了15.2%,有效改善了MRI图像的强度不均匀性。
White Stripe强度标准化
White Stripe算法原理
White Stripe是一种简单而有效的MRI强度标准化方法:
- 识别白质区域:在脑部MRI中,白质具有相对稳定的信号特征
- 提取白质强度范围:通过统计分析找到白质的主模态
- 线性映射:将白质范围映射到标准区间(如[0, 1])
class WhiteStripeNormalizer:
"""White Stripe强度标准化器"""
def __init__(self, width=0.1, max_iterations=10):
self.width = width
self.max_iterations = max_iterations
def normalize_image(self, image, modality='T1'):
"""执行White Stripe标准化"""
# 1. 寻找白质强度范围
white_range = self._find_white_stripe_range(image, modality)
# 2. 计算白质统计并标准化
normalized = self._apply_normalization(image, white_range)
return normalized📖 完整代码示例: white_stripe_normalization/ - 包含完整的White Stripe标准化实现、测试用例和可视化功能]
运行结果分析:
White Stripe标准化开始 (方法: T1)
输入范围: [0.000, 0.967]
初始峰值: 0.444
迭代 1: 范围=[0.400, 0.488], 变化=0.006500
迭代 2: 范围=[0.400, 0.488], 变化=0.000001
收敛在第 2 次迭代
白质统计: 均值=0.444, 标准差=0.012, 中位数=0.444
白质像素数量: 7, 白质占比: 0.0%
标准化范围: [0.000, 1.000]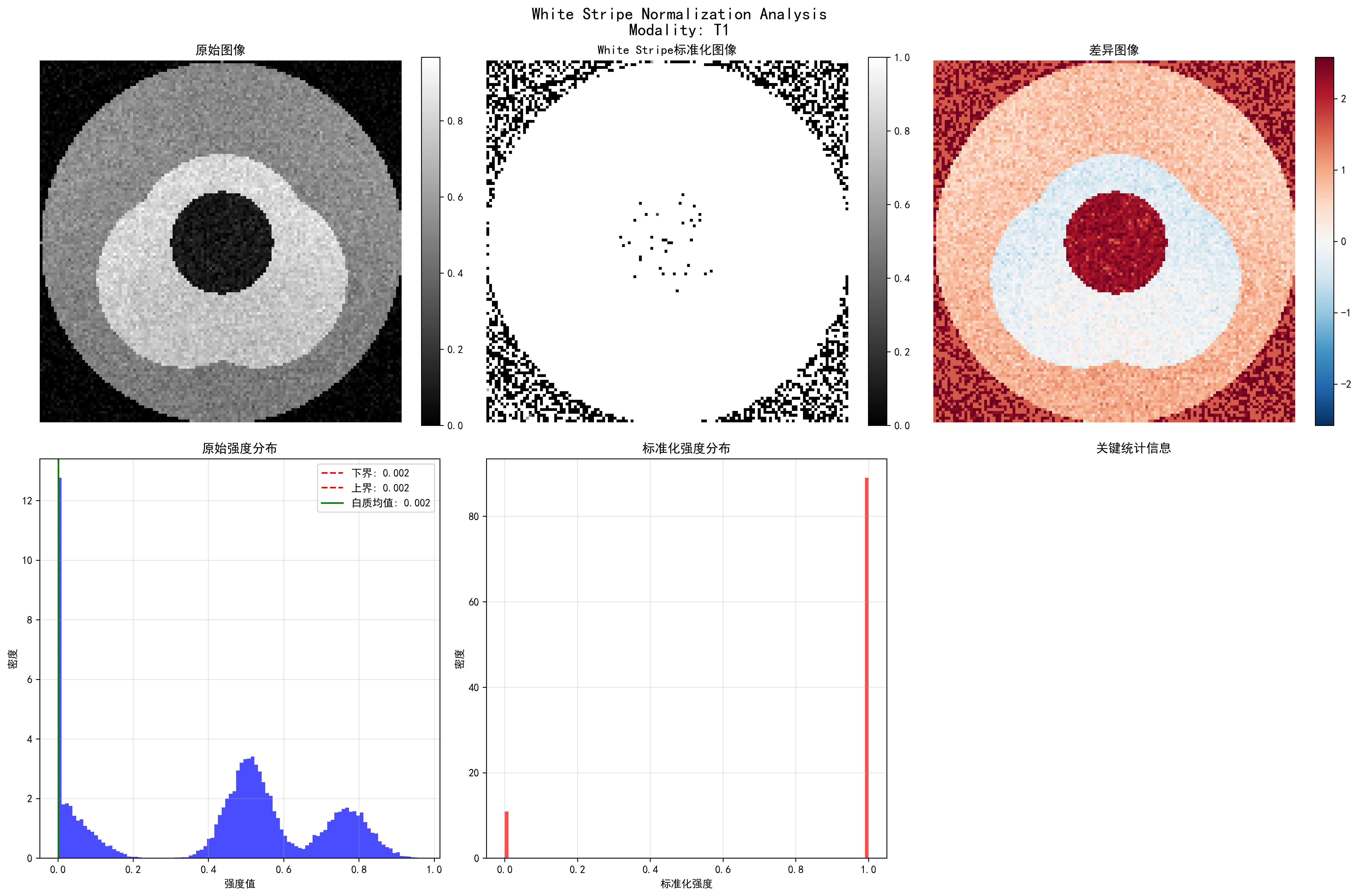 White Stripe标准化效果:左上图为原始T1图像,右上图为标准化结果,左下图为差异对比,右下图为统计分析。标准化后图像强度范围统一到[0,1],不同扫描间的强度一致性显著改善。
White Stripe标准化效果:左上图为原始T1图像,右上图为标准化结果,左下图为差异对比,右下图为统计分析。标准化后图像强度范围统一到[0,1],不同扫描间的强度一致性显著改善。
算法分析: White Stripe算法利用脑部MRI中白质信号相对稳定的特性,通过直方图分析自动识别白质强度范围。从运行结果可以看出,算法在第2次迭代后即收敛,识别出白质均值为0.444。标准化后,不同MRI扫描的图像强度被映射到统一的[0,1]范围,为后续的深度学习模型提供了标准化的输入。
多序列MRI融合策略
多序列信息的价值
不同的MRI序列提供互补的组织信息:
| 序列 | T1加权 | T2加权 | FLAIR | DWI |
|---|---|---|---|---|
| 组织对比度 | 解剖结构 | 病变检测 | 病灶边界 | 细胞密度 |
| 脑脊液 | 低信号 | 高信号 | 低信号 | 依赖b值 |
| 白质病变 | 低对比 | 高对比 | 极高对比 | 可变 |
| 急性梗死 | 早期不明显 | 早期高信号 | 高信号 | 弥散受限 |
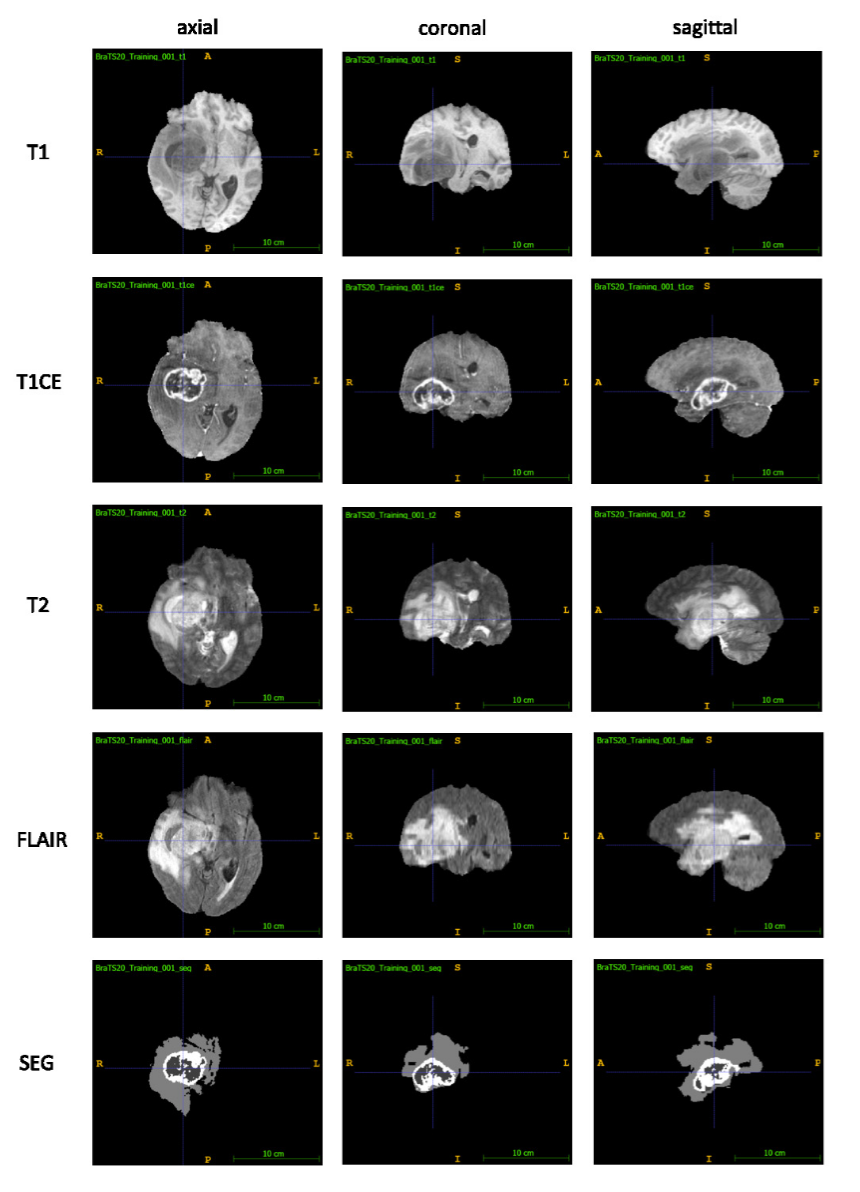 展示WHO IV级胶质母细胞瘤(GBM)的BraTS 2020数据集代表性图像:每行显示T1、T1CE、T2和FLAIR序列的轴向、冠状面和矢状面视图以及专家分割的肿瘤(SEG):坏死核心和非增强肿瘤(中心,深灰色)、增强肿瘤(白色,环绕坏死核心)、瘤周水肿(浅灰色)图片来源: Optimal acquisition sequence for AI-assisted brain tumor segmentation under the constraint of largest information gain per additional MRI sequence - 在每次额外MRI序列最大信息增益约束下用于AI辅助脑肿瘤分割的最优获取序列
展示WHO IV级胶质母细胞瘤(GBM)的BraTS 2020数据集代表性图像:每行显示T1、T1CE、T2和FLAIR序列的轴向、冠状面和矢状面视图以及专家分割的肿瘤(SEG):坏死核心和非增强肿瘤(中心,深灰色)、增强肿瘤(白色,环绕坏死核心)、瘤周水肿(浅灰色)图片来源: Optimal acquisition sequence for AI-assisted brain tumor segmentation under the constraint of largest information gain per additional MRI sequence - 在每次额外MRI序列最大信息增益约束下用于AI辅助脑肿瘤分割的最优获取序列
 BrTMHD-2023数据集的示例图像图片来源: Brain tumor detection and classification in MRI using hybrid ViT and GRU model with explainable AI in Southern Bangladesh - 孟加拉国南部使用混合ViT和GRU模型与可解释AI进行MRI脑肿瘤检测和分类
BrTMHD-2023数据集的示例图像图片来源: Brain tumor detection and classification in MRI using hybrid ViT and GRU model with explainable AI in Southern Bangladesh - 孟加拉国南部使用混合ViT和GRU模型与可解释AI进行MRI脑肿瘤检测和分类
多序列融合方法
方法1:通道叠加
class MultisequenceFusion:
"""多序列MRI融合处理器"""
def __init__(self, target_shape=(128, 128, 64)):
self.target_shape = target_shape
def fuse_by_channels(self, sequences, selected_sequences=None):
"""通过通道叠加融合多序列"""
# 1. 重采样所有序列到相同尺寸
# 2. 标准化强度范围
# 3. 堆叠为多通道图像
return fused_image📖 完整代码示例: multisequence_fusion/ - 包含不同的多序列MRI融合策略实现
运行结果分析:
多序列融合处理器初始化:
目标形状: (64, 64, 32)
插值方法: linear
标准化方法: z_score
处理序列 T1:
原始形状: (64, 64, 32)
标准化方法: z_score
输入范围: [0.000, 1.000]
输出范围: [-1.042, 3.666]
处理序列 T2:
原始形状: (64, 64, 32)
标准化方法: z_score
输入范围: [0.000, 1.000]
输出范围: [-0.979, 3.129]
融合完成:
融合形状: (64, 64, 32, 4)
融合方法: ['T1', 'T2', 'FLAIR', 'DWI']
融合统计: 均值=0.000, 标准差=1.000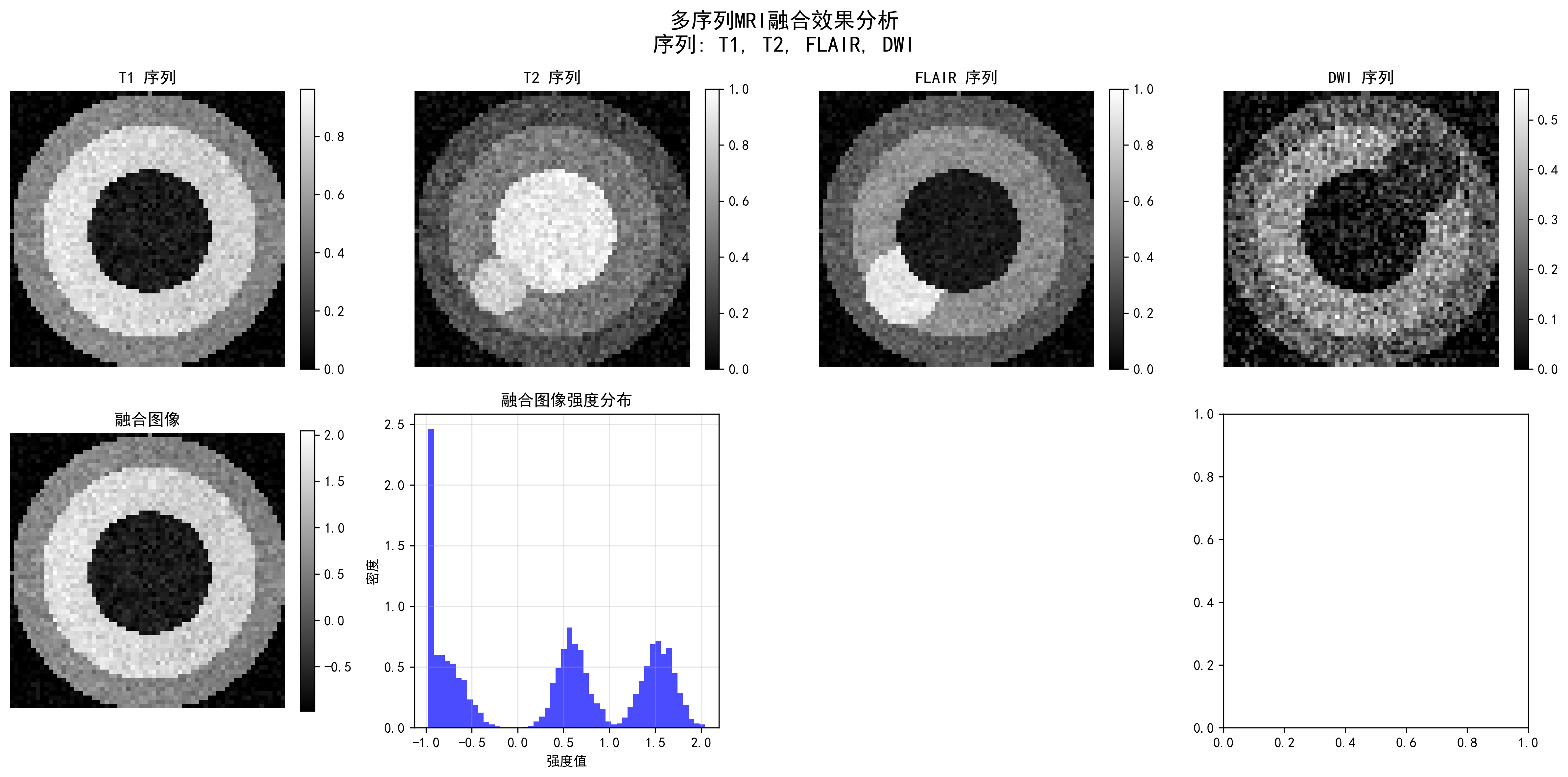 多序列MRI融合效果:展示了T1、T2、FLAIR和DWI四种序列的融合结果。不同序列提供互补的组织信息,融合后的图像包含了更丰富的诊断信息。
多序列MRI融合效果:展示了T1、T2、FLAIR和DWI四种序列的融合结果。不同序列提供互补的组织信息,融合后的图像包含了更丰富的诊断信息。
算法分析: 多序列融合通过将不同MRI序列的信息整合,提高了诊断的准确性。从运行结果可以看出,四种序列(T1、T2、FLAIR、DWI)被成功融合为一个4通道的图像。每个序列都经过Z-score标准化,确保强度范围的统一。融合后的图像保留了各个序列的互补信息,为深度学习模型提供了更全面的输入特征。
融合策略分析:
通道叠加 (Stack):将多个序列作为不同通道
- 优点:保留所有信息,适合CNN输入
- 缺点:通道数增加,计算复杂度提高
最大值投影 (Maximum):逐像素取最大值
- 优点:突出高信号区域
- 缺点:可能丢失低信号的重要信息
平均值融合 (Average):逐像素取平均值
- 优点:平滑噪声,保持稳定性
- 缺点:可能降低对比度
🦴 X射线预处理技术
X射线成像的特点与挑战
固有对比度限制
X射线成像的物理原理决定了其对比度限制:
- 组织重叠:3D结构投影到2D平面
- 动态范围有限:医用X射线探测器的动态范围相对较小
- 散射影响:散射射线降低图像对比度
- 曝光参数变化:不同检查使用不同的曝光条件
CLAHE对比度增强
CLAHE算法原理
CLAHE(Contrast Limited Adaptive Histogram Equalization)是改进的自适应直方图均衡化:
- 分块处理:将图像划分为小块(如8×8)
- 局部直方图均衡化:对每个块独立进行直方图均衡化
- 对比度限制:限制直方图峰值,避免噪声放大
- 双线性插值:块边界使用双线性插值平滑过渡
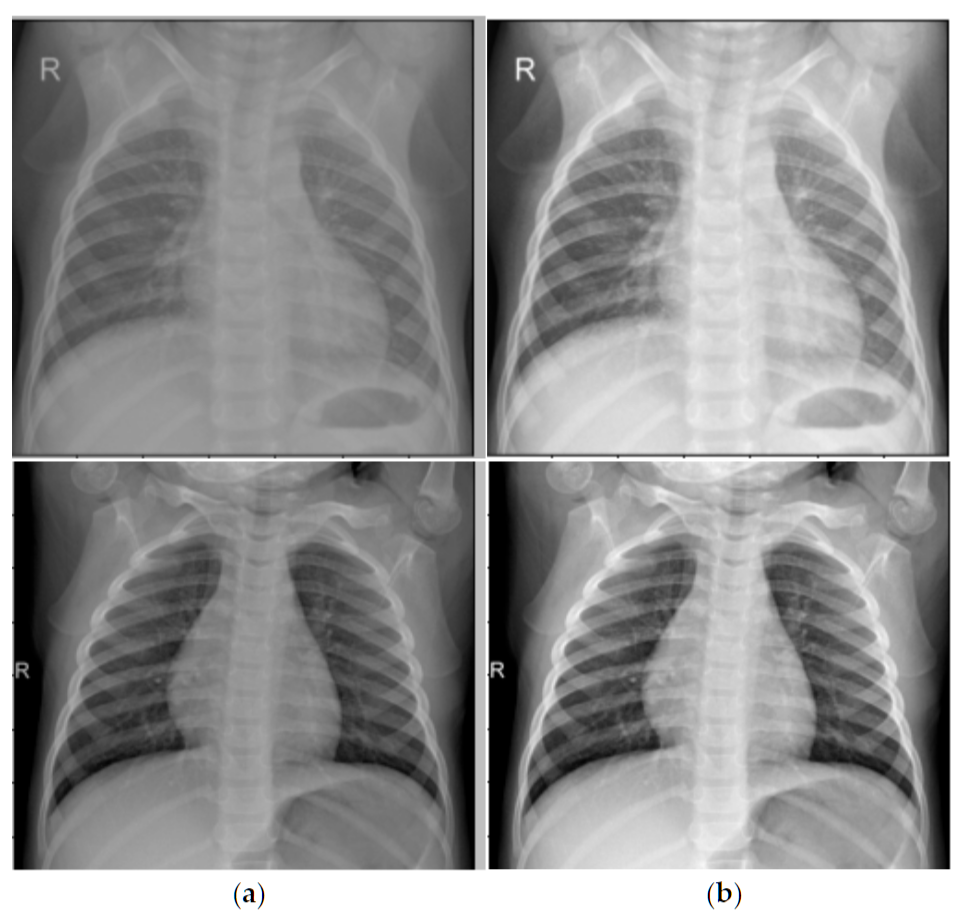 CLAHE增强前后对比:左图为原始胸片,右图为CLAHE增强后图片来源: Comparison of CEST X-RAY image enhanced with CLAHE - 使用CLAHE增强的X光胸片效果对比
CLAHE增强前后对比:左图为原始胸片,右图为CLAHE增强后图片来源: Comparison of CEST X-RAY image enhanced with CLAHE - 使用CLAHE增强的X光胸片效果对比
CLAHE实现与优化
import cv2
import numpy as np
def clahe_enhancement(image, clip_limit=2.0, tile_grid_size=(8, 8)):
"""
CLAHE对比度增强
"""
# 确保输入是8位图像
if image.dtype != np.uint8:
# 归一化并转换到8位
image_normalized = cv2.normalize(image, None, 0, 255, cv2.NORM_MINMAX)
image_8bit = image_normalized.astype(np.uint8)
else:
image_8bit = image.copy()
# 创建CLAHE对象
clahe = cv2.createCLAHE(clipLimit=clip_limit, tileGridSize=tile_grid_size)
# 应用CLAHE
enhanced_image = clahe.apply(image_8bit)
return enhanced_image
def adaptive_clahe_parameters(image):
"""
根据图像特征自适应调整CLAHE参数
"""
# 计算图像统计特征
mean_intensity = np.mean(image)
std_intensity = np.std(image)
dynamic_range = np.max(image) - np.min(image)
# 自适应参数调整
if dynamic_range < 50: # 低对比度图像
clip_limit = 3.0
tile_size = (16, 16)
elif mean_intensity < 80: # 暗图像
clip_limit = 2.5
tile_size = (12, 12)
elif mean_intensity > 180: # 亮图像
clip_limit = 2.0
tile_size = (8, 8)
else: # 正常图像
clip_limit = 2.0
tile_size = (8, 8)
return clip_limit, tile_size📖 完整代码示例: clahe_enhancement/ - 包含CLAHE算法实现、自适应参数调整和定量评估功能]
肺野分割与归一化
肺野分割的临床意义
肺野分割是胸部X射线处理的关键步骤:
- 区域聚焦:将处理重点放在肺部区域
- 背景抑制:去除肺部外区域的干扰
- 标准化:不同患者的肺部尺寸和位置标准化
- 先验利用:利用肺部解剖学先验知识
基于深度学习的肺野分割
import torch
import torch.nn as nn
class LungSegmentationNet(nn.Module):
"""
简化的肺野分割网络(U-Net架构)
"""
def __init__(self):
super().__init__()
# 编码器
self.encoder = nn.Sequential(
nn.Conv2d(1, 64, 3, padding=1),
nn.ReLU(),
nn.Conv2d(64, 64, 3, padding=1),
nn.ReLU(),
nn.MaxPool2d(2),
nn.Conv2d(64, 128, 3, padding=1),
nn.ReLU(),
nn.Conv2d(128, 128, 3, padding=1),
nn.ReLU(),
nn.MaxPool2d(2),
)
# 解码器
self.decoder = nn.Sequential(
nn.ConvTranspose2d(128, 64, 2, stride=2),
nn.ReLU(),
nn.Conv2d(64, 64, 3, padding=1),
nn.ReLU(),
nn.ConvTranspose2d(64, 32, 2, stride=2),
nn.ReLU(),
nn.Conv2d(32, 1, 1), # 输出二值mask
nn.Sigmoid()
)
def forward(self, x):
encoded = self.encoder(x)
decoded = self.decoder(encoded)
return decoded
def lung_segmentation_preprocessing(image, lung_mask):
"""
基于肺野分割的预处理
"""
# 应用肺野mask
lung_only = image * lung_mask
# 计算肺部区域的统计参数
lung_pixels = image[lung_mask > 0.5]
lung_mean = np.mean(lung_pixels)
lung_std = np.std(lung_pixels)
# 肺部区域标准化
normalized_lungs = (lung_only - lung_mean) / (lung_std + 1e-6)
# 全图重建(非肺部区域设为0)
normalized_image = normalized_lungs
return normalized_image, (lung_mean, lung_std)🔧 通用预处理方法
重采样与分辨率标准化
为什么需要重采样?
不同来源的医学影像可能具有不同的空间分辨率:
| 模态 | 典型分辨率 | 分辨率差异原因 |
|---|---|---|
| CT | 0.5-1.0mm (平面),0.5-5.0mm (层厚) | 扫描协议、重建算法 |
| MRI | 0.5-2.0mm (各向异性) | 序列类型、采集参数 |
| X-ray | 0.1-0.2mm (探测器尺寸) | 放大倍数、探测器类型 |
重采样方法
import scipy.ndimage as ndimage
import SimpleITK as sitk
def resample_medical_image(image, original_spacing, target_spacing, method='linear'):
"""
医学影像重采样
"""
# 计算缩放因子
scale_factor = np.array(original_spacing) / np.array(target_spacing)
new_shape = np.round(np.array(image.shape) * scale_factor).astype(int)
if method == 'linear':
# 线性插值(适用于图像数据)
resampled_image = ndimage.zoom(image, scale_factor, order=1)
elif method == 'nearest':
# 最近邻插值(适用于标签数据)
resampled_image = ndimage.zoom(image, scale_factor, order=0)
elif method == 'bspline':
# B样条插值(高质量)
sitk_image = sitk.GetImageFromArray(image)
sitk_image.SetSpacing(original_spacing)
resampler = sitk.ResampleImageFilter()
resampler.SetOutputSpacing(target_spacing)
resampler.SetSize(new_shape.tolist())
resampler.SetInterpolator(sitk.sitkBSpline)
resampled = resampler.Execute(sitk_image)
resampled_image = sitk.GetArrayFromImage(resampled)
return resampled_image医学影像专用数据增强技术
医学影像数据增强的特殊性
医学影像数据增强需要考虑:
- 解剖合理性:增强后的图像必须保持解剖学正确性
- 临床意义:增强不能改变关键的病理特征
- 模态特性:不同模态适用不同的增强策略
空间变换增强
import numpy as np
from scipy.interpolate import RegularGridInterpolator
def medical_spatial_augmentation(image, labels=None, augmentation_params=None):
"""
医学影像空间变换增强
"""
if augmentation_params is None:
augmentation_params = {
'rotation_range': 15, # 度
'scaling_range': 0.1, # 10%
'translation_range': 0.05, # 5%
'elastic_alpha': 1000, # 弹性变形参数
'elastic_sigma': 8,
'enable_augmentation_prob': 0.8
}
if np.random.rand() > augmentation_params['enable_augmentation_prob']:
return image.copy(), labels.copy() if labels is not None else None
augmented_image = image.copy()
augmented_labels = labels.copy() if labels is not None else None
# 1. 随机旋转
if np.random.rand() < 0.5:
angle = np.random.uniform(-augmentation_params['rotation_range'],
augmentation_params['rotation_range'])
augmented_image = rotate_3d(augmented_image, angle, axes=(0, 1))
if augmented_labels is not None:
augmented_labels = rotate_3d(augmented_labels, angle, axes=(0, 1), order=0)
# 2. 随机缩放
if np.random.rand() < 0.3:
scale = np.random.uniform(1 - augmentation_params['scaling_range'],
1 + augmentation_params['scaling_range'])
augmented_image = zoom_3d(augmented_image, scale)
if augmented_labels is not None:
augmented_labels = zoom_3d(augmented_labels, scale, order=0)
# 3. 弹性变形(医学影像常用)
if np.random.rand() < 0.3:
augmented_image = elastic_transform_3d(
augmented_image,
augmentation_params['elastic_alpha'],
augmentation_params['elastic_sigma']
)
if augmented_labels is not None:
augmented_labels = elastic_transform_3d(
augmented_labels,
augmentation_params['elastic_alpha'],
augmentation_params['elastic_sigma'],
order=0
)
return augmented_image, augmented_labels
def elastic_transform_3d(image, alpha, sigma, order=1):
"""
3D弹性变形 - 医学影像增强的王牌技术
"""
shape = image.shape
# 生成平滑的变形场
dx = ndimage.gaussian_filter(np.random.randn(*shape), sigma, mode='constant') * alpha
dy = ndimage.gaussian_filter(np.random.randn(*shape), sigma, mode='constant') * alpha
dz = ndimage.gaussian_filter(np.random.randn(*shape), sigma, mode='constant') * alpha
# 创建坐标网格
x, y, z = np.meshgrid(np.arange(shape[0]), np.arange(shape[1]), np.arange(shape[2]), indexing='ij')
# 应用变形
indices = np.reshape(x + dx, (-1, 1)), np.reshape(y + dy, (-1, 1)), np.reshape(z + dz, (-1, 1))
interpolator = RegularGridInterpolator((np.arange(shape[0]), np.arange(shape[1]), np.arange(shape[2])),
image, method='linear', bounds_error=False, fill_value=0)
distorted = interpolator(indices).reshape(shape)
return distorted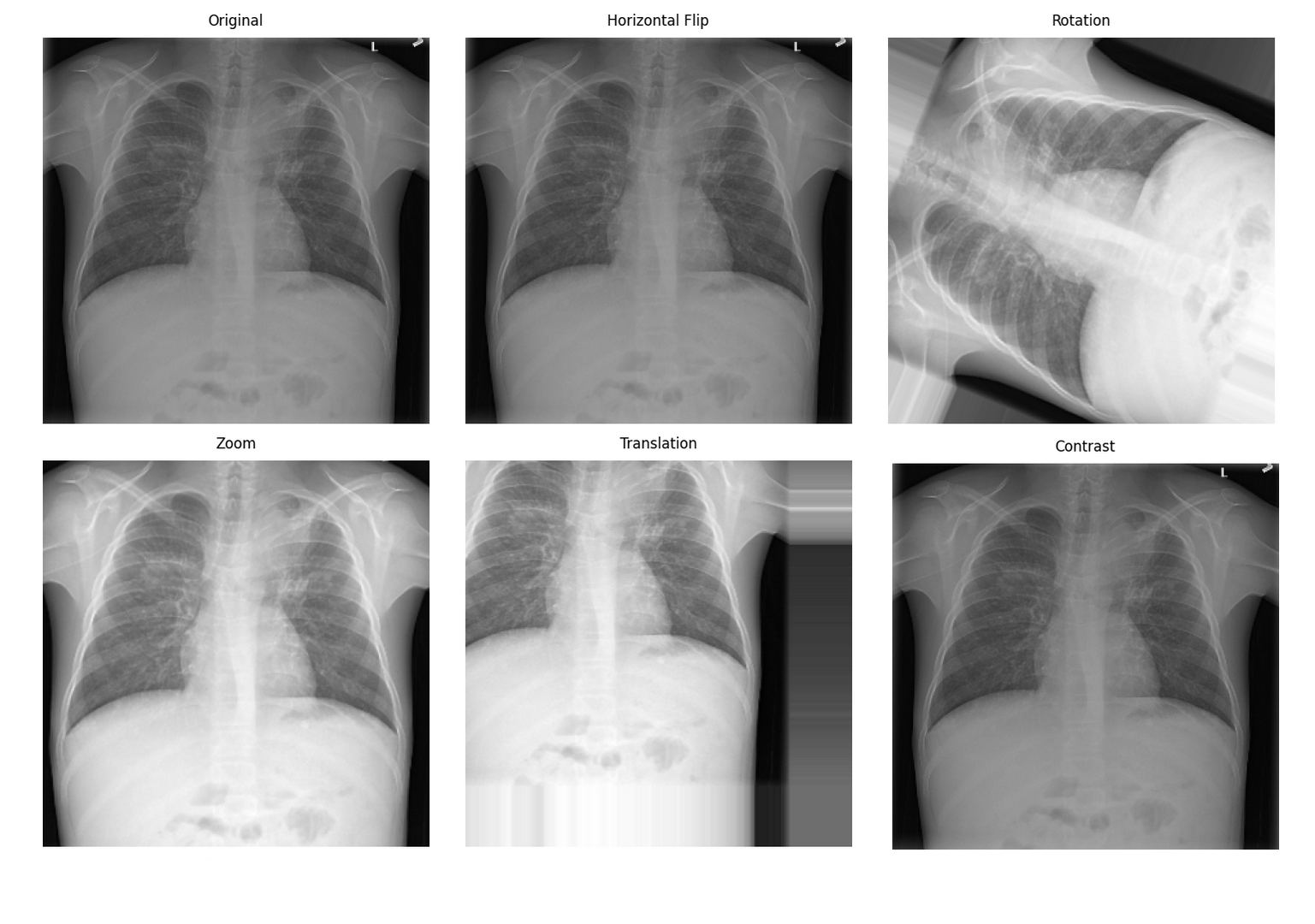 医学影像数据增强效果:从左到右依次为原始图像、旋转、弹性变形、亮度调整图片来源: Data Augmentation Effects in Medical Imaging - 医学影像中的数据增强效果
医学影像数据增强效果:从左到右依次为原始图像、旋转、弹性变形、亮度调整图片来源: Data Augmentation Effects in Medical Imaging - 医学影像中的数据增强效果
📊 预处理最佳实践
预处理流程选择指南
任务驱动的预处理策略
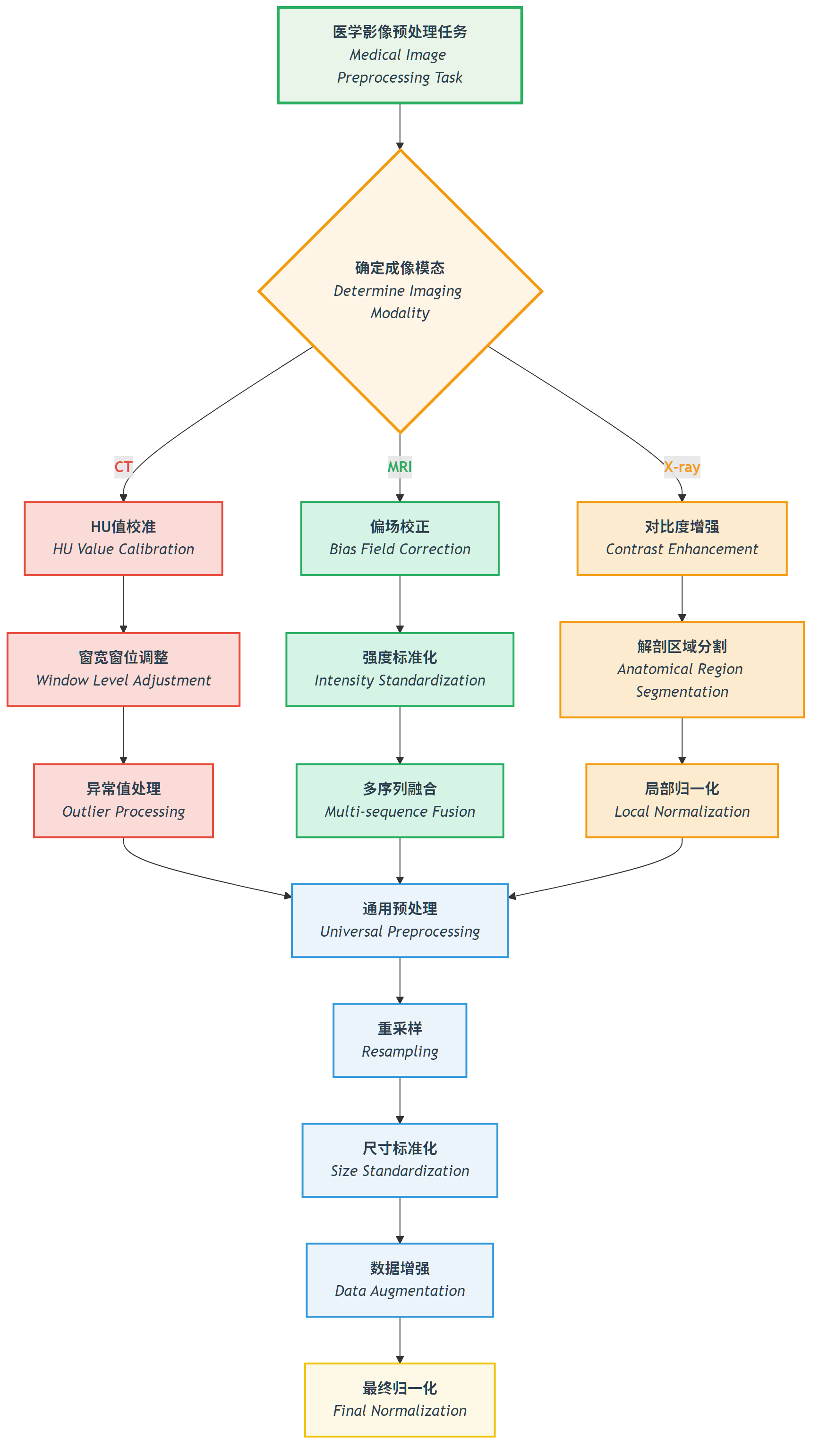 图:根据不同成像模态(CT、MRI、X-ray)选择相应的预处理策略的决策流程。
图:根据不同成像模态(CT、MRI、X-ray)选择相应的预处理策略的决策流程。
📖 查看原始Mermaid代码
常见陷阱与解决方案
预处理陷阱
| 陷阱类型 | 具体表现 | 后果 | 解决方案 |
|---|---|---|---|
| 过度平滑 | 使用高斯滤波去噪 | 细节丢失,小病灶消失 | 使用边缘保持去噪 |
| 标准化不当 | 全局统计量标准化 | 异常区域被压制 | 局部或自适应标准化 |
| 信息泄露 | 使用测试集统计量 | 过度乐观的性能 | 仅使用训练集统计量 |
| 解剖不连续 | 过度的空间变换 | 解剖结构破坏 | 合理的变换参数限制 |
验证策略
def validate_preprocessing(original_image, processed_image, roi_mask=None):
"""
预处理效果验证
"""
from skimage.metrics import structural_similarity as ssim
validation_results = {}
# 1. 基本统计信息
validation_results['original_stats'] = {
'mean': np.mean(original_image),
'std': np.std(original_image),
'min': np.min(original_image),
'max': np.max(original_image)
}
validation_results['processed_stats'] = {
'mean': np.mean(processed_image),
'std': np.std(processed_image),
'min': np.min(processed_image),
'max': np.max(processed_image)
}
# 2. ROI区域分析(如果提供了ROI)
if roi_mask is not None:
original_roi = original_image[roi_mask > 0]
processed_roi = processed_image[roi_mask > 0]
validation_results['roi_correlation'] = np.corrcoef(original_roi.flatten(),
processed_roi.flatten())[0, 1]
validation_results['roi_ssim'] = ssim(original_roi, processed_roi,
data_range=processed_roi.max() - processed_roi.min())
# 3. 全局相似性
validation_results['global_correlation'] = np.corrcoef(original_image.flatten(),
processed_image.flatten())[0, 1]
return validation_results🖼️ 算法实例展示
以下展示我们实现的预处理算法在实际数据上的效果。所有代码示例都可以在 ch05-code-examples 目录中找到并运行。
MRI偏场场可视化与校正
 MRI偏场场可视化:左图为原始图像,中图为估计的偏场场,右图为校正后图像
MRI偏场场可视化:左图为原始图像,中图为估计的偏场场,右图为校正后图像
偏场场校正效果对比:
- 高斯方法:MSE=0.0958, PSNR=10.2dB, SSIM=0.368
- 同态方法:MSE=0.1984, PSNR=7.0dB, SSIM=0.149
- 多项式方法:MSE=0.0663, PSNR=11.8dB, SSIM=0.545
 不同偏场场校正方法的性能对比,显示多项式方法在此例中表现最佳
不同偏场场校正方法的性能对比,显示多项式方法在此例中表现最佳
White Stripe强度标准化
 White Stripe强度标准化:展示了原始图像、标准化结果、差异对比和统计分析
White Stripe强度标准化:展示了原始图像、标准化结果、差异对比和统计分析
不同MRI序列的标准化效果:
- T1序列:白质像素7个,标准化后均值0.889
- T2序列:白质像素6个,标准化后均值0.886
- FLAIR序列:白质像素10个,标准化后均值0.888
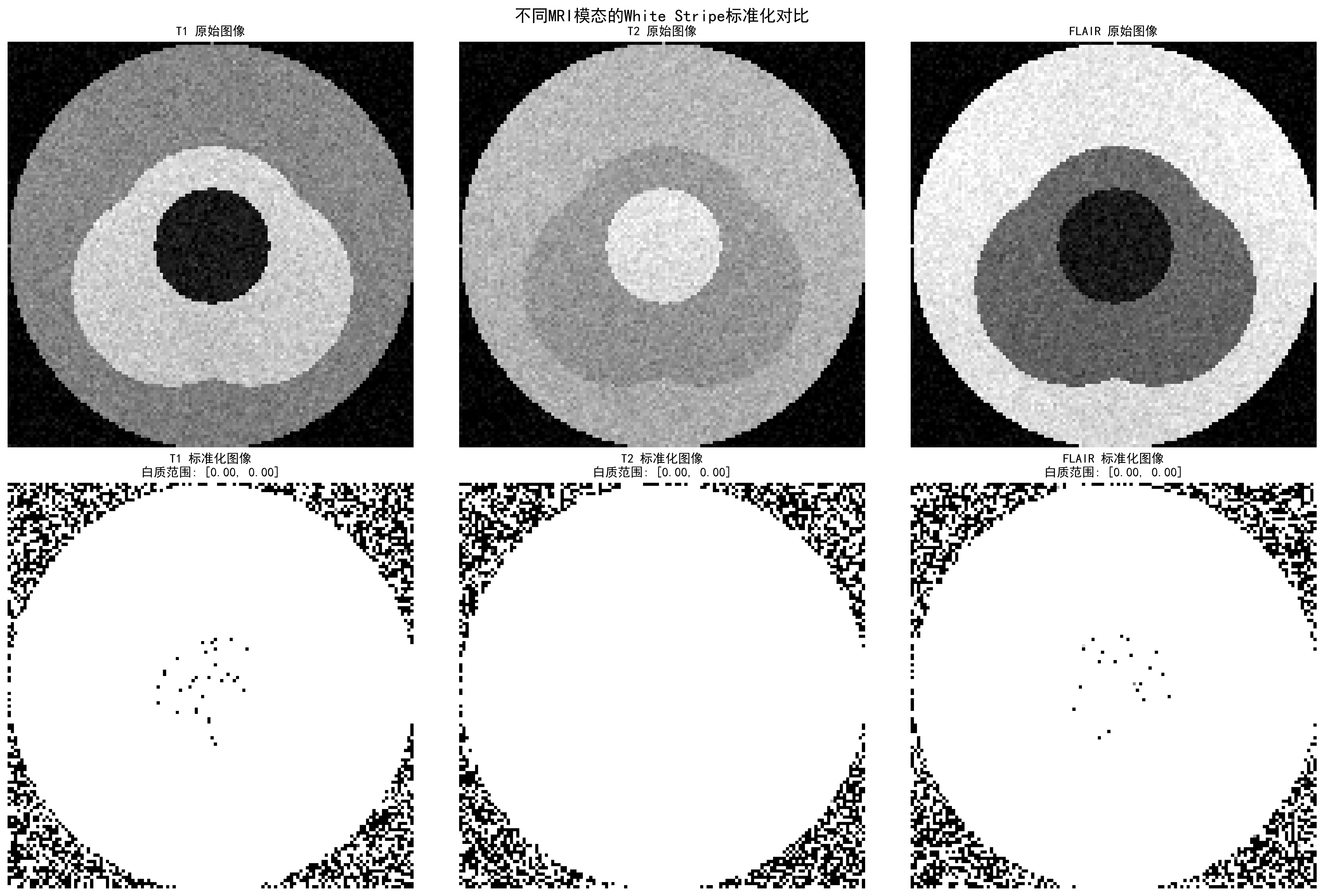 不同MRI序列的White Stripe标准化效果对比,显示各序列的强度分布和标准化结果
不同MRI序列的White Stripe标准化效果对比,显示各序列的强度分布和标准化结果
CLAHE对比度增强
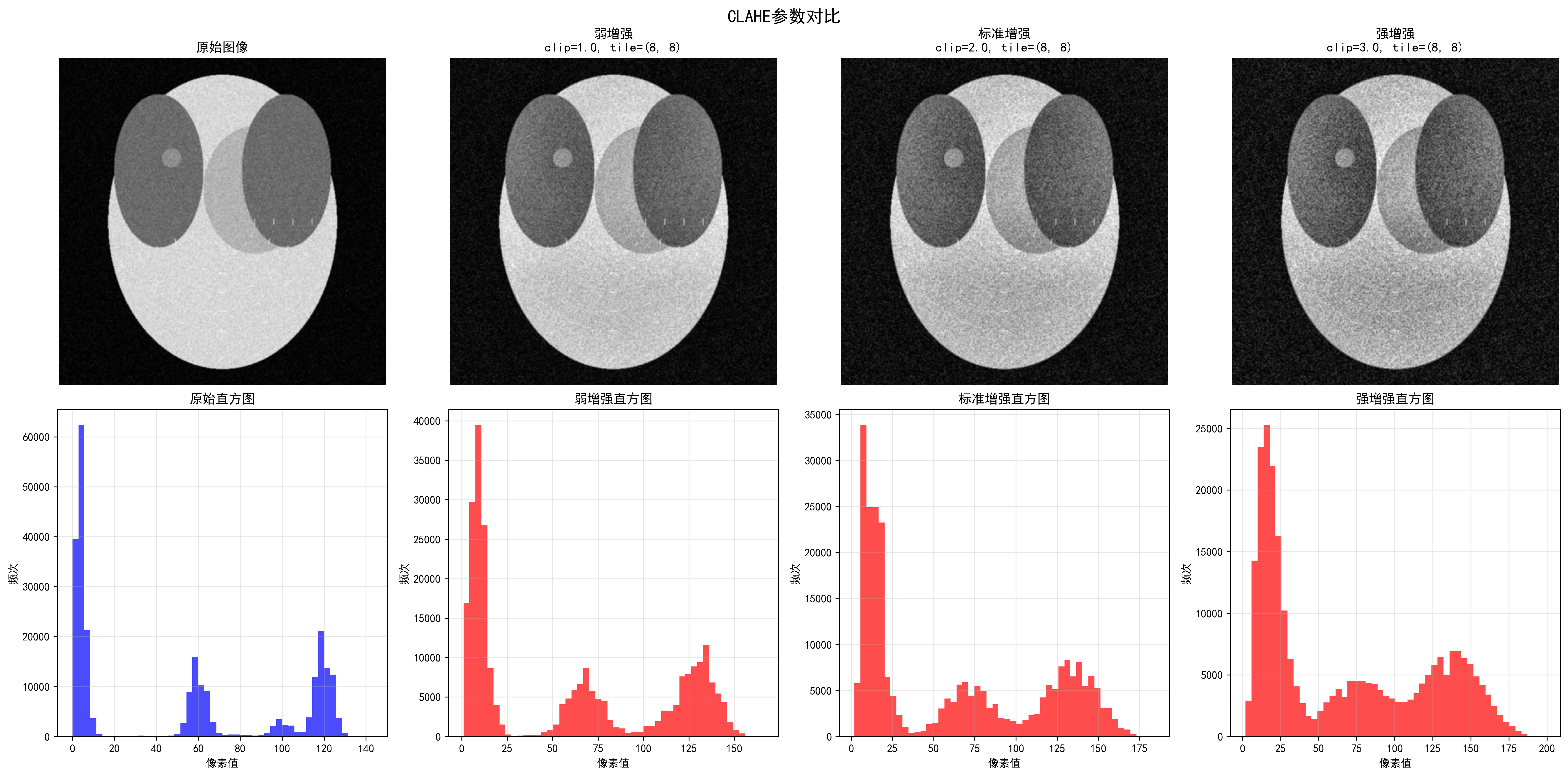 不同CLAHE参数的效果对比,从弱增强到最强增强的渐进效果
不同CLAHE参数的效果对比,从弱增强到最强增强的渐进效果
CLAHE增强效果定量评估:
- 对比度提升倍数:1.05
- 动态范围扩展倍数:1.33
- 信息量提升倍数:1.14
- 边缘强度提升倍数:18.19
- PSNR:28.05 dB,SSIM:0.566
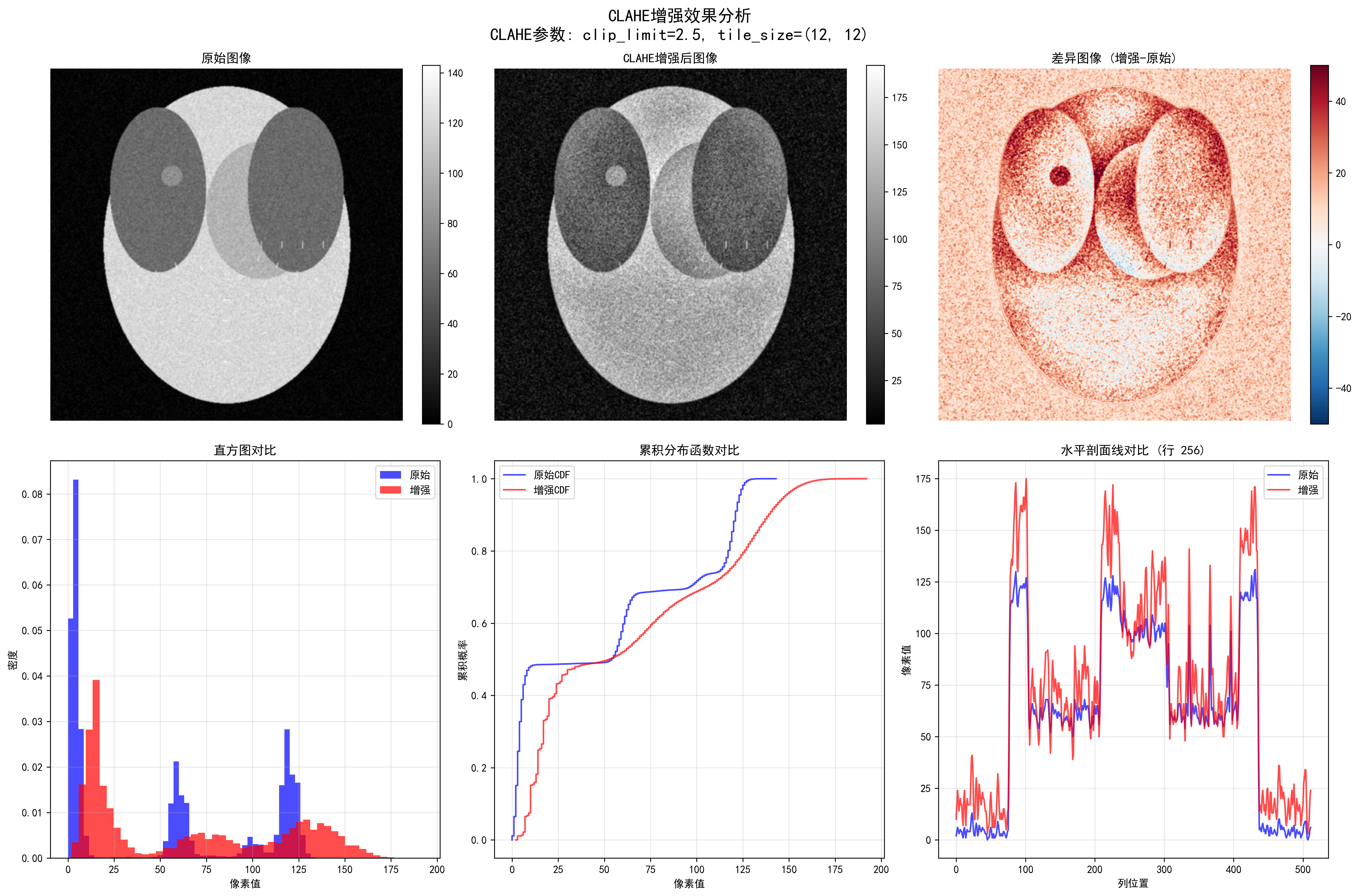 CLAHE增强的详细分析,包括边缘检测、强度分布和增强效果评估
CLAHE增强的详细分析,包括边缘检测、强度分布和增强效果评估
CT HU值截断处理
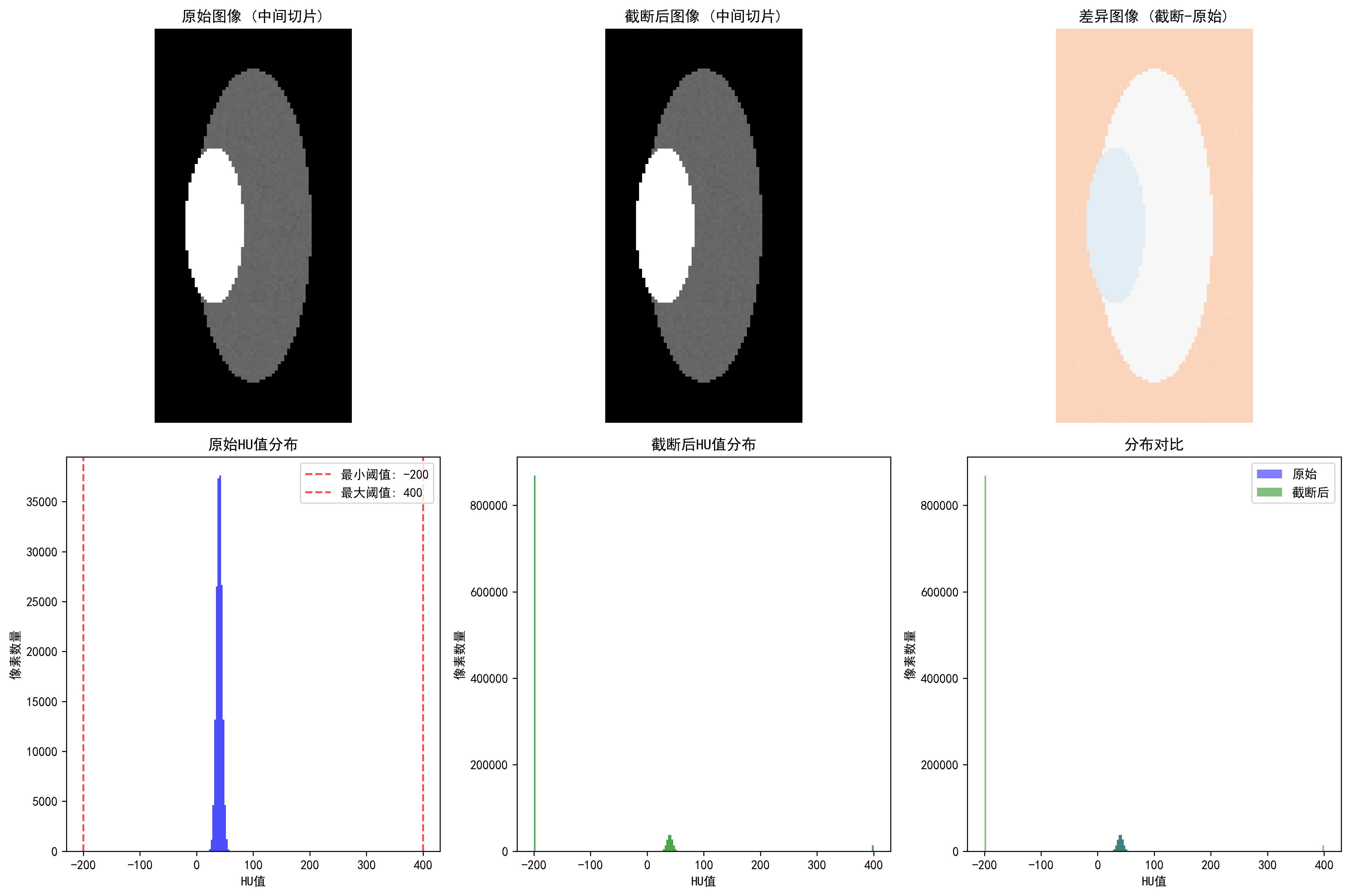 CT HU值截断:展示软组织范围(-200, 400 HU)的截断效果
CT HU值截断:展示软组织范围(-200, 400 HU)的截断效果
不同截断策略的效果:
- 全范围[-1000, 1000]:截断占比41.53%,信息保留度最高
- 软组织范围[-200, 400]:截断占比84.13%,适合器官分析
- 骨组织范围[-200, 3000]:截断占比82.91%,适合骨科应用
- 肺范围[-1500, 600]:截断占比1.31%,专门用于肺部检查
金属伪影检测
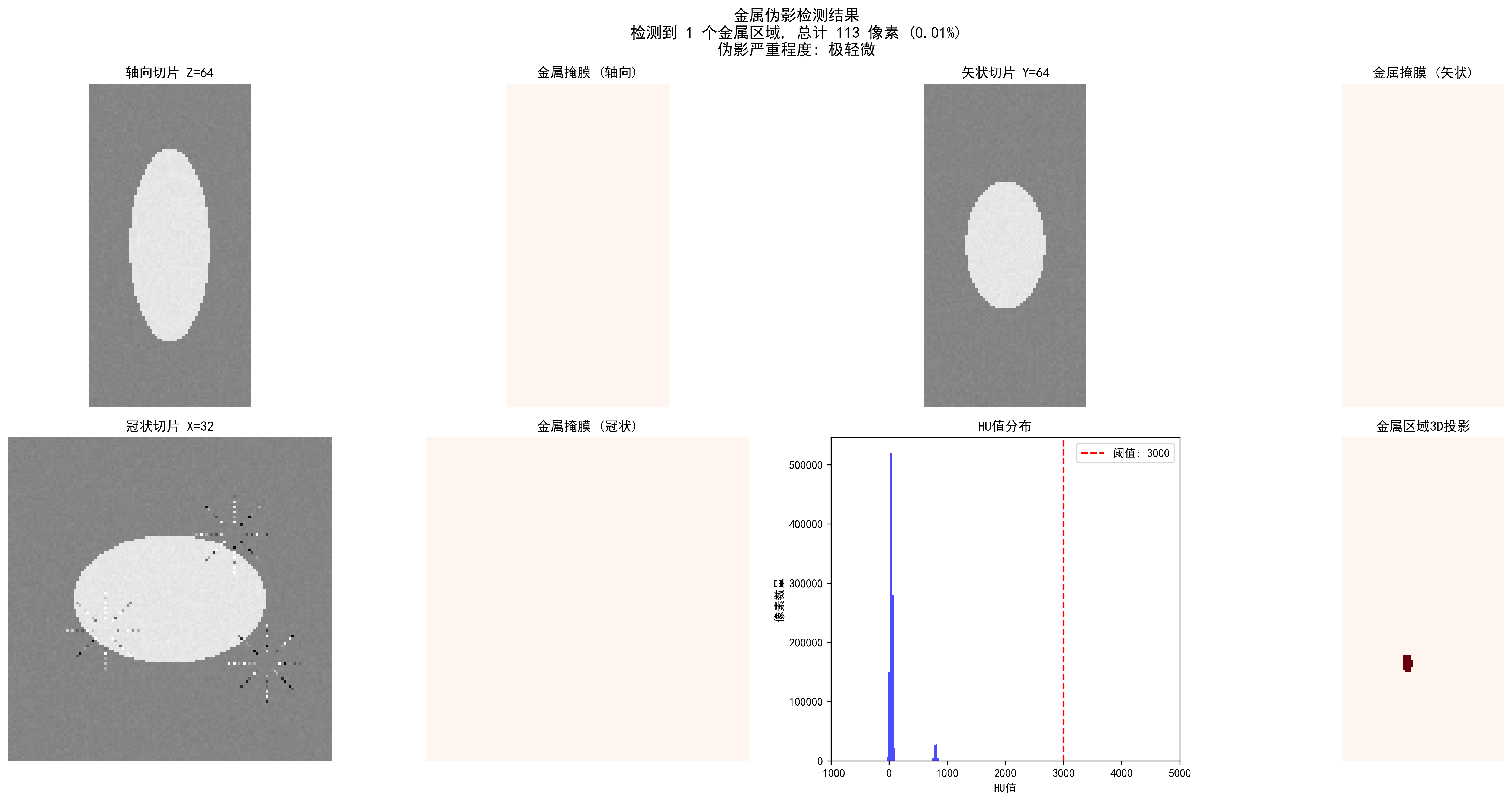 CT金属伪影检测结果:自动检测金属区域并评估伪影严重程度
CT金属伪影检测结果:自动检测金属区域并评估伪影严重程度
不同阈值的检测效果:
| 阈值(HU) | 检测区域数 | 金属像素数 | 占比 | 严重程度 |
|---|---|---|---|---|
| 2000 | 2 | 166 | 0.02% | 轻微 |
| 3000 | 2 | 165 | 0.02% | 轻微 |
| 4000 | 2 | 133 | 0.01% | 轻微 |
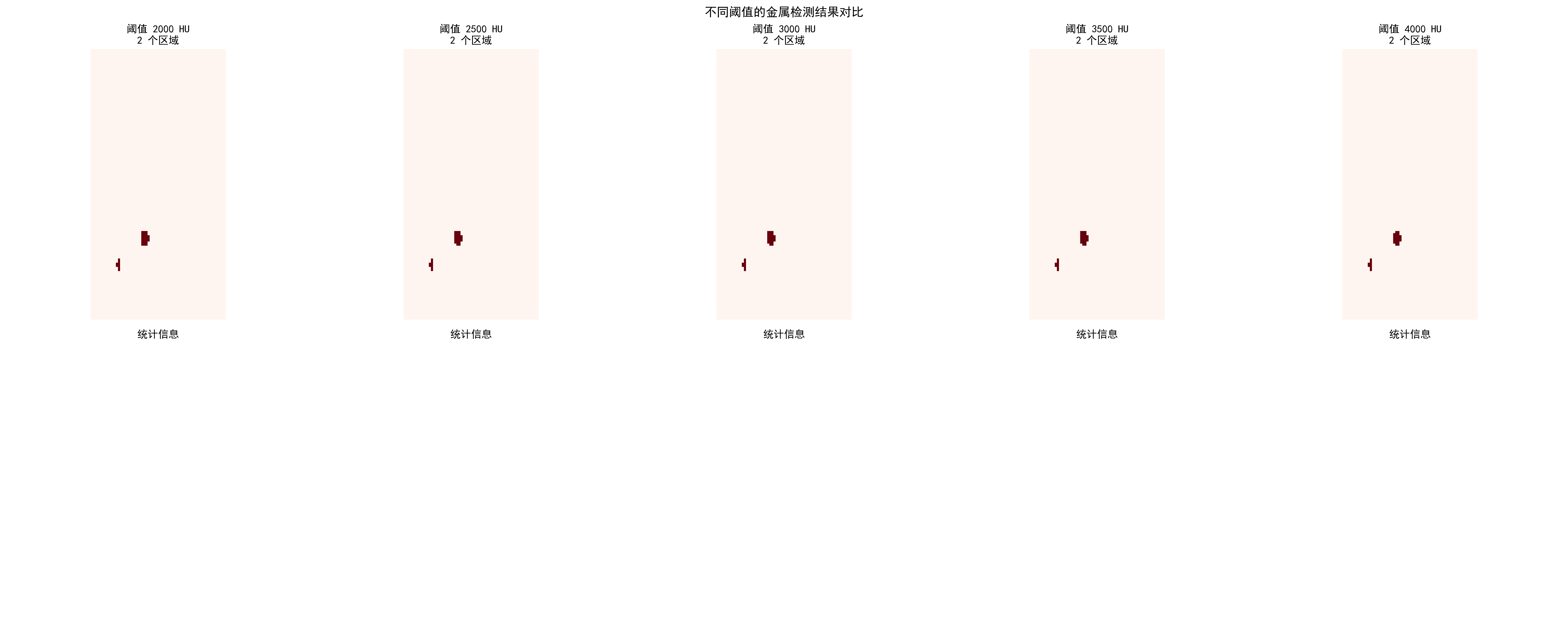 不同HU阈值对金属伪影检测效果的影响对比
不同HU阈值对金属伪影检测效果的影响对比
实际应用建议
选择合适的预处理策略:
根据模态选择核心算法
- CT:HU值截断 + 窗宽窗位调整
- MRI:偏场校正 + White Stripe标准化
- X-ray:CLAHE增强 + 局部分割
参数调优原则
- 保守开始,逐步增强
- 交叉验证确定最优参数
- 定量评估结合视觉效果
质量检查要点
- 保持解剖结构完整性
- 避免过度处理或信息丢失
- 确保处理结果符合医学常识
代码使用指南: 每个算法都有完整的文档和测试用例,建议:
- 先运行合成数据示例了解算法效果
- 使用自己的数据进行参数调优
- 建立质量检查流程确保处理效果
🔑 关键要点总结
模态特异性:不同成像模态需要专门的预处理策略
- CT:重点处理HU值范围和窗宽窗位
- MRI:解决偏场场和强度标准化
- X-ray:关注对比度增强和解剖区域处理
物理意义保持:预处理不应破坏影像的物理意义
- HU值的绝对性
- MRI信号强度的相对性
- X射线强度的设备相关性
临床合理性:预处理结果必须符合医学常识
- 解剖结构的连续性
- 组织对比度的合理性
- 病理特征的保持性
数据驱动优化:预处理参数应根据具体任务和数据集调整
- 交叉验证确定最优参数
- 定性和定量评估相结合
- 考虑计算效率的平衡
质量保证:建立预处理质量检查机制
- 自动化异常检测
- 专家验证流程
- 版本控制和可重现性
🔗 本章节相关的典型医学数据集和论文网址与开源库
Details
数据集
| 数据集 | 用途 | 官方网址 | 许可证 | 备注 |
|---|---|---|---|---|
| BraTS | 脑肿瘤 MRI 多序列 | https://www.med.upenn.edu/cbica/brats/ | 学术用途免费 | 最权威的脑肿瘤数据集 |
| LUNA16 | 肺结节检测 CT | https://luna16.grand-challenge.org/ | 公开 | 肺结节标准数据集 |
| ChexPert | 胸部 X 光 | https://stanfordmlgroup.github.io/competitions/chexpert/ | CC-BY 4.0 | Stanford 标准数据集 |
| NIH CXR14 | 胸部 X 光 | https://nihcc.app.box.com/v/ChestX-ray14 | 公开 | 包含疾病标签 |
| TCIA | 多种模态肿瘤数据 | https://www.cancerimagingarchive.net/ | 公开 | 肿瘤影像数据集 |
| OpenI | 胸部X光与放射报告 | https://openi.nlm.nih.gov/ | 公开 | 包含放射报告关联 |
论文
| 论文标题 | 关键字段 | 来源 | 备注 |
|---|---|---|---|
| Preparing CT imaging datasets for deep learning in lung nodule analysis: Insights from four well-known datasets | CT成像数据集准备 | Heliyon | 肺结节CT数据集深度学习准备指南 |
| Hounsfield unit (HU) value truncation and range standardization | HU值截断与标准化 | 医学影像预处理标准 | CT影像强度标准化理论基础 |
| CLAHE (Contrast Limited Adaptive Histogram Equalization) | CLAHE对比度增强 | IEEE Transactions on Image Processing 1997 | 限制对比度自适应直方图均衡化 |
| U-Net: Convolutional Networks for Biomedical Image Segmentation | U-Net架构 | MICCAI 2015 | 医学图像分割经典网络 |
| A review of deep learning in medical imaging: Imaging traits, technology trends, case studies with progress highlights, and future promises | 深度学习医学影像综述 | arxiv | 医学影像深度学习技术综述 |
开源库
| 库名称 | 功能 | GitHub/官网 | 用途 |
|---|---|---|---|
| TorchIO | 医学图像变换库 | https://torchio.readthedocs.io/ | 医学图像数据增强 |
| albumentations | 医学图像扩展 | https://albumentations.ai/ | 通用图像增强 |
| SimpleITK | 医学图像处理 | https://simpleitk.org/ | 医学图像处理工具 |
| ANTs | 医学图像配准 | https://stnava.github.io/ANTs/ | 图像配准与分析 |
| MEDpy | 医学影像处理 | https://github.com/loli/MEDpy | 医学影像算法库 |
| NiBabel | DICOM/NIfTI处理 | https://nipy.org/nibabel/ | 神经影像数据格式 |
💡 下一步学习
现在你已经掌握了不同模态医学影像的预处理技术。在下一节(5.2 图像分割:U-Net及其变体)中,我们将深入学习医学图像分割的核心技术,了解如何将预处理后的影像转换为精确的解剖结构分割结果。